MOE
(統合計算化学システム)
MOE(Molecular Operating Environment)は、低分子、ペプチド、タンパク質、抗体、および核酸などの多様な創薬モダリティに対応した分子設計とモデリングソフトウェアです。多彩なアプリケーションと豊富なデータコンテンツを搭載しており、計算科学者だけでなく、合成研究者、バイオロジスト、X線結晶構造解析者などの研究活動を強力に支援します。 グラフィックス、コマンドライン、ウェブアプリケーション、ワークフローの各種使用モードと柔軟なカスタマイズにより、ユーザーの研究目的にあわせて最適な分子モデリング環境を構築できます。
tab:{
title:{機能}
3Dグラフィックス
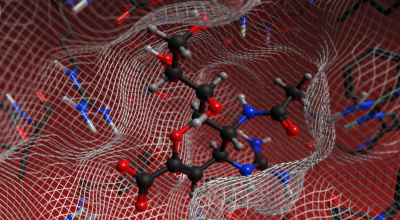
- ユーザーフレンドリーなグラフィカルユーザーインターフェース
- 非結合相互作用の可視化
- 高品質な画像と動画
- GPU アクセラレーション 3D ステレオ グラフィックス
- 複合仮想現実と3Dプリント
Structure-Based Design
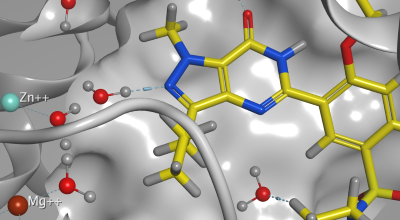
- 活性部位の検出と解析
- ポケット内でのリガンド設計
- タンパク質-リガンド相互作用図
- ウォーターサイトとエネルギーの予測
- 誘導フィットドッキング
- フラグメントのリンク、伸長、置換
抗体と生物学的製剤の設計
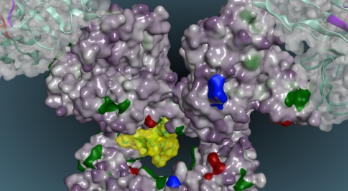
- 立体構造に基づくタンパク質工学
- 開発可能性と問題を引き起こす可能性(liability)の評価
- 親和性、安定性、溶解度の最適化
- ハイスループット抗体モデリング
- バーチャルライブラリの生成
- タンパク質ータンパク質ドッキングとエピトープマッピング
MOEsaic - SAR解析
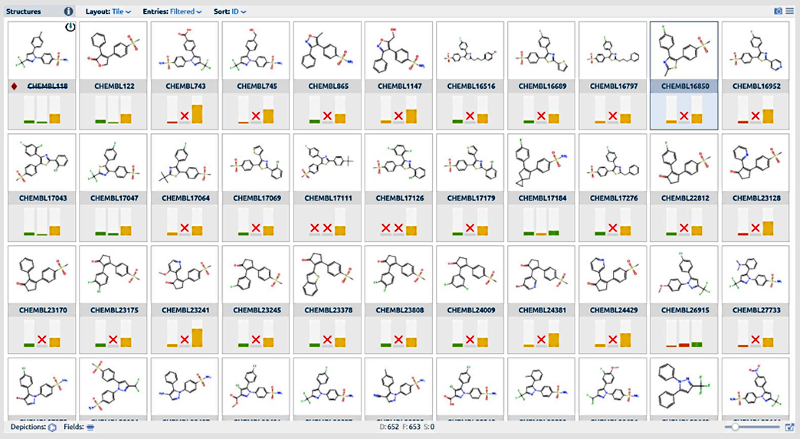
- SARとSPRの可視化
- Free-Wilson法による化合物の提案
- Matched Molecular Pairs(MMP)解析
- 置換基解析とプロファイリング
- 部分構造および類似構造検索
- 新規仮想化合物の設計
Ligand-Based Design
- 配座の生成とクラスタリング
- 低分子のアラインメントと重ね合わせ
- ファーマコフォア解析とスクリーニング
- コンビナトリアルライブラリーの生成
タンパク質、核酸モデリング
- タンパク質とその相互作用残基、および特徴的分子表面の可視化
- アミノ酸配列からのタンパク質立体構造予測
- DNA/RNAモデルの構築
- 突然変異体と側鎖配座の探索
- 分子動力学シミュレーション
- ループ/リンカー探索とサンプリング
- タンパク質-タンパク質ドッキング
バーチャルスクリーニング
- 3Dファーマコフォアスクリーニング
- 特徴と形状による束縛条件
- 低分子ドッキング
- 2Dおよび3Dフィンガープリントスクリーニング
- 高速な配座解析データベース構築
- 化学反応ベースのライブラリー設計
Fragment-Based Design
- 母核置換
- フラグメントの連結および伸長
- メディシナルケミストリー変換
- リガンド・ハイブリダイゼーション(BREED)
- フラグメント・ライブラリー
構造バイオインフォマティクス
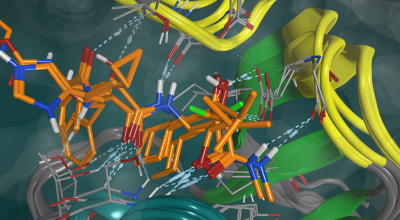
- マルチ配列、および構造アラインメント
- 3D構造ベースのアミノ酸配列アノテーション
- タンパク質ファミリーデータベースの構築と検索
- 構造データマイニング
分子シミュレーション
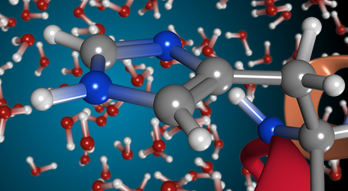
- 分子力学計算、および分子動力学計算
- 分子系の前処理の自動化
- 自由エネルギー計算
- 複数分子のフレキシブルアラインメント
- 配座解析 ーLowModeMDー
- 2面角のスキャンと解析
- QMベースのMNR、IR、およびVCDスペクトル
ペプチドモデリング
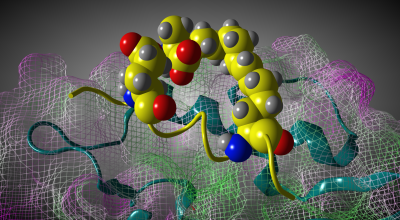
- 大環状、および線形ペプチド
- ペプチドータンパク質相互作用の同定
- 配座解析
- 非天然ペプチドライブラリの網羅的生成
- 立体構造に基づくペプチド設計
- ペプチドのプロパティの最適化
- ペプチドドッキング
構造生物学
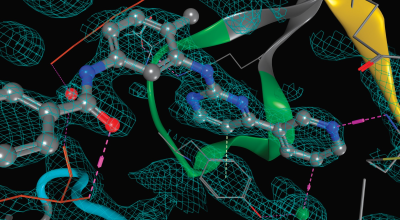
- 電子密度、および差異マップの描画
- 結晶格子と結晶構造における原子接触の可視化
- 水分子の位置の予測
- 電子密度誘導ドッキング
- タンパク質ファミリーのアラインメントデータベース
- 分子置換のためのホモロジーモデリング
- タンパク質立体構造の妥当性の検証
ケモインフォマティクスとQSAR
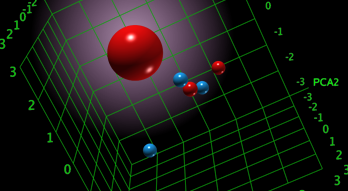
- 400以上の2Dおよび3D分子記述子
- pKaの予測と水素付加状態の網羅的生成
- 線形QSAR/QSPRモデル構築
- ベイジアン分類モデル
- 化合物の類似性、ダイバーシティ、およびクラスタリング
カスタマイズと開発環境
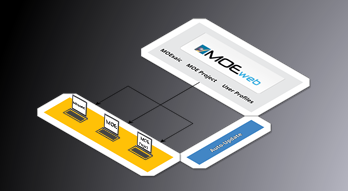
- ラップトップ - クラスターマシン - クラウドコンピューティング - ワークフロー
- Windows - Linux - macOS
- 統合型開発環境(SVL - Scientific Vector Language)
- サードパーティソフトウェアの統合
- カスタムアプリケーション
- ウェブ統合、ウェブサービス、およびAPI
PSILO - タンパク質立体構造データベース
- ウェブブラウザインターフェース - 3D分子構造表示
- プロジェクトデータベースの自動キュレーション
- 3D相互作用検索と統計解析
- ポケット類似性検索
- タンパク質立体構造の重ね合わせ
開発元について
title:{サポート}
トレーニング
トレーニングの詳細は、こちらをご覧ください。
title:{SVLプログラムのダウンロード}
CCG社のSVL ExchangeからMOEを使用してアドオンのSVLプログラムをダウンロードできます。
title:{ドキュメント}
MOE2024.06 システム環境解説書
MOE 2024.06のシステム構成、動作環境、各種データベースについて解説
MOE システム環境解説書
MOE2024.06 インストール手順書
MOEのインストール手順を詳細に解説
Windows版
MacOS版
Linux版
アーカイブからのインストール
インストールDVDからインストールする場合の注意事項
【重要】データファイルの結合方法を解説(インストールDVDからインストールする際はこの操作が必要)
【重要】データファイルの準備
ライセンス更新手順書
新しく発行されたライセンスファイルの更新手順を簡易的に解説
ライセンスファイル更新手順書
ライセンスサーバーのリプレース等で、ライセンスサーバーよりライセンスサービスを削除する操作を解説
ライセンスサービスの削除
MOE 2024.0601のリリースに伴うインストールファイル名の変更について
MOE 2024.0601ではインストールファイル名が変更されました。各種手順書を本PDFを参照して読み替えてください。
MOE 2024.06マイナーバージョンアップに伴うインストール用ファイル名変更について
title:{論文一覧}
MOEを使用した論文の一覧です。研究の参考にご利用下さい。
(現在弊社で把握しているものであり、MOEを使用した論文の全てを掲載しているわけではありません。 論文を出版された方、一覧に掲載されていない論文をご存知の方は、こちらまでお知らせいただけましたら幸いです。)
また、CCG社のMOE Citationsページもご参照ください。
MOEを使用した論文
Binary quantitative structure – activity relationship (QSAR) analysis of estrogen receptor ligands.J. Chem. Inf. Comput. Sci., 1999, 39, 164-168.
Mol. Inf. 2010, 29, 243-249.
J. Comput. Aided. Chem., 2010, 11, 56-61.
Clin. Infect. Dis., 2005, 41, 243-251.
当社製MOEアドオンプログラムASEDockを使用した論文
2013
https://www.riken.jp/press/2024/20240719_4/index.html
205 – 211.
当社製MOEアドオンプログラムAutoQSARを使用した論文
2047-2056.
title:{化合物カタログ}
MOE版化合物カタログお問い合わせ先
MOEは独自のデータベース機能により大量の化合物データも容易に取り扱えます。この機能を活かし試薬供給会社と提携して、MOE版化合物カタログを発行しています。これらカタログに登録されている延べ数百万件の化合物データを新薬候補化合物のin silico での探索にご利用頂けます。
MOE版化合物カタログでは、化合物の2次元構造に加え、物理化学的特性やフィンガープリント情報の付加を行っています。これにより、特性による絞り込みや類似化合物検索にカタログデータをそのまま使用できます。またMOE-QFSS用のインデックスファイルも収録しておりますので、MOE-QFSSを用いて部分構造/類似構造の超高速検索を実行することもできます。 MOE版化合物カタログは以下のご連絡先にお問い合わせ下さい。
ナミキ商事株式会社 ファインケミカル事業部
TEL:03-3354-4026
FAX:03-3352-2196
近年、創薬研究の効率化が多くの場面で叫ばれる中、インシリコスクリーニングの技術を用い優良な化合物を見出す作業は益々重要性を増しております。 弊社では引き続き世界各国のサプライヤーと連携しスクリーニング用化合物のデータを収集し、供給性の高い独自のデータベースの構築を行っております(全750万化合物を収載)。 データベースをご希望の場合は無償にてご提供致しますので弊社までお問合せを頂けますと幸いです。
キシダ化学株式会社 技術支援部 ケミカルグループ
TEL:03-5625-5595
FAX:03-5625-5596
探索研究においてハイスループットスクリーニング(HTS)が普及したことに伴い、膨大な数のスクリーニングサンプルが求められるようになりました。弊社はこうしたニーズにお応えすべく、全世界から低分子化合物を取り揃え、随時新たな化合物を追加しデータベースを更新しております。 これらのスクリーニング用化合物を、お客様のご要望に応じたフォーマットで提供してきておりますので、ご興味お持ちのお客様は弊社まで御連絡ください。
title:{動作環境}
動作環境
MOE 2024.06は以下のハードウェア・OSに対応しています。
| アーキテクチャ | オペレーティングシステム(OS) | グラフィックス |
|---|---|---|
| intel | Windows 11/10 64bit | OpenGL |
| intel | Linux 64bit(RHEL 7, Ubuntu 12.04, Debian 8.0, Fedora 16, OpenSUSE 12.1 相当以上)(glibc 2.14以上) | OpenGL, X11 |
| intel | macOS 10.12以上 | OpenGL, X11 |
| Apple Silicon (M1以上) | macOS 11以上 | OpenGL, X11 |
使用モード
| 名称 | 概要 | 消費トークン | 説明 |
|---|---|---|---|
| MOE | グラフィックモード | 3 |
MOEのグラフィック(GUI)モードです。 通常のユーザまたは、アプリケーション開発者が使用します。 MOEの全ての機能が使用できます。また、操作性が最も優れたモードです。 |
| MOE/batch | コマンドモード | 1 |
MOEのノングラフィックモードです。 長時間の計算をサーバ機で実行する場合に使用します。 グラフィックモードと比較して計算速度が1割程度高速になります。MOEをサードパーティー製品に組み込んで使用することができます。 |
| MOE/web | Webサーバ、SOAPサーバモード | 1以上 |
MOEのWebサーバ兼SOAPサーバモードです。 MOEの特定の機能を、Webブラウザから簡単に使用できます。また、SOAPに対応したワークフローツールからhttpプロトコルを通してMOEの機能を使用できます。 |
| MOE/smp | 分散処理モード | 1以上 | 大量のデータを複数マシンに分割処理する計算モードです。 MOEの並列計算対応アプリケーションを並列計算で実行する場合に使用します。MOE/smpは、MOEがサポートしている全てのOSを混在させたヘテロクラスタを作成することも可能です。 |
title:{パンフレット}
MOEの各種パンフレットのダウンロード
MOE全体パンフレット
MOE パンフレット (5.5MB)
MOE Brochure (English, 2MB)
MOE機能別パンフレット
MOE 2024.06新機能パンフレット (0.6MB)
MOEsaic (0.8MB)
核酸モデリング(0.4MB)
抗体モデリング (0.7MB)
タンパク質変異体解析 (0.5MB)
ペプチド解析 (0.3MB)
Solvent Analysis (0.3MB)
MOEカスタムアプリケーション
Bio-MOE(バイオ医薬品開発アプリケーション) (0.6MB)
MOE-QFSS(化合物ライブラリー超高速検索) (0.6MB)
MOE-ASEDock(ドッキングシミュレーション)(0.6MB)
MOE-Extensions for KNIME (MOE KNIME I/F) (0.7MB)
}:tab





