MOE - 抗体と生物学的製剤の設計
tab:{
title:{概要}
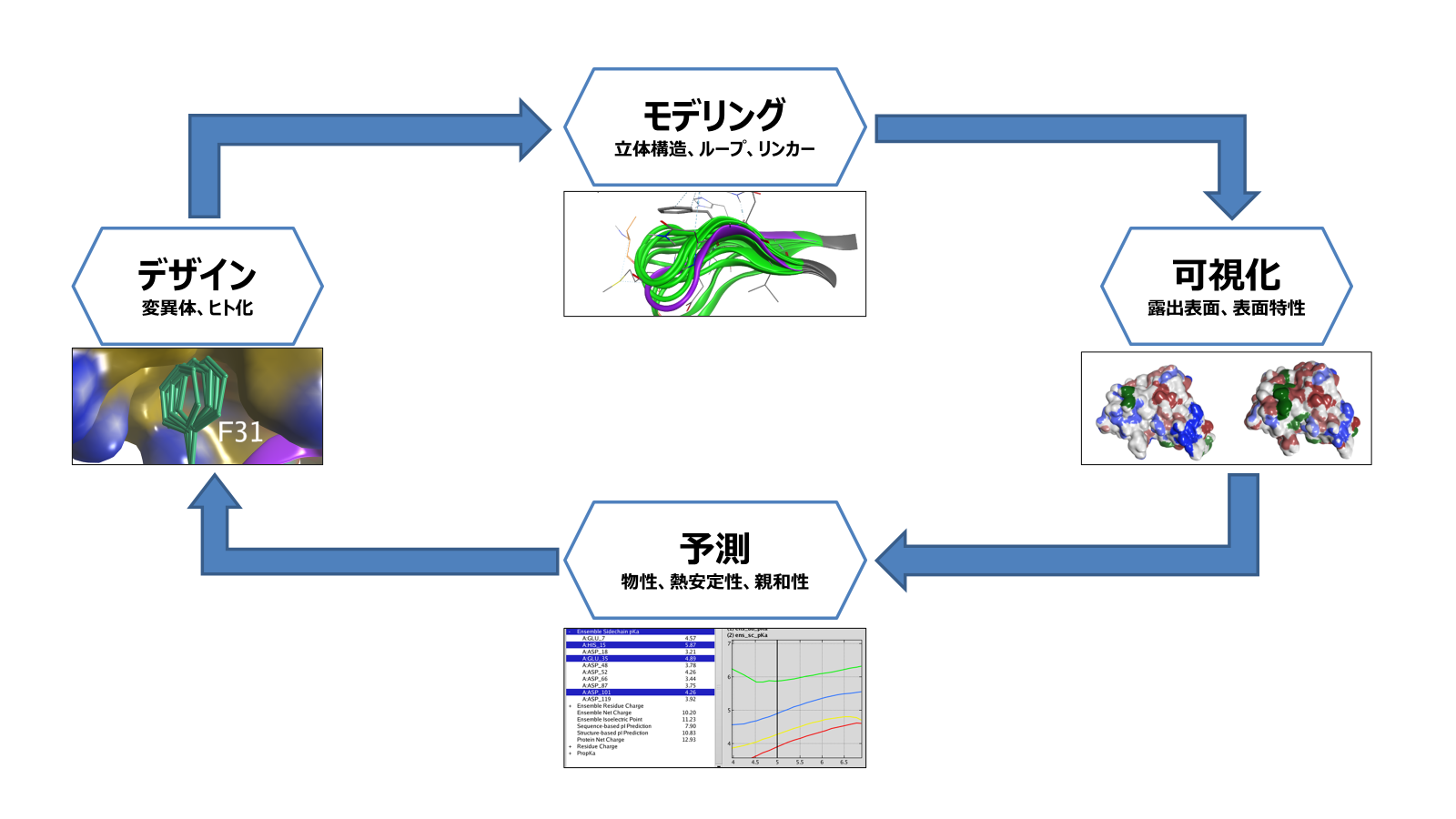
近年、生物学的製剤は、さまざまな疾患の治療においてその重要性がますます高まっています。中でも抗体医薬品は、多くの実績がある生物学的製剤の一つです。 抗体医薬品の開発には、インシリコ技術が不可欠であり、抗体の立体構造予測、ヒト化、抗原との相互作用解析、エピトープマッピング、デザイン計算、物性予測など、幅広い解析が求められます。 MOEでは、これらすべての解析を包括的に実施でき、抗体医薬品をはじめとする生物学的製剤の開発を強力に支援します。
title:{抗体モデリング}
抗体モデリング
簡単な操作で適切な抗体モデリングを行えます。抗体配列を入力することで、自動的に相補性決定領域(CDR)とフレームワーク(FR)領域を認識し、領域ごとにテンプレート構造の検索を行うことで、抗体のホモロジーモデルを構築します。それぞれの領域について配列相同性や構造品質などを考慮したテンプレート候補が提案されます。Fv、Fab、F(ab’)、rIgG、IgG、VHH、VLL、一本鎖抗体(scFv)、二重特異性抗体(bispecific抗体)などのフォーマットを指定できます。ヒト由来のテンプレートを選択すれば、抗体のヒト化モデリングも可能です。複数配列を入力とした連続モデリングにも対応しています
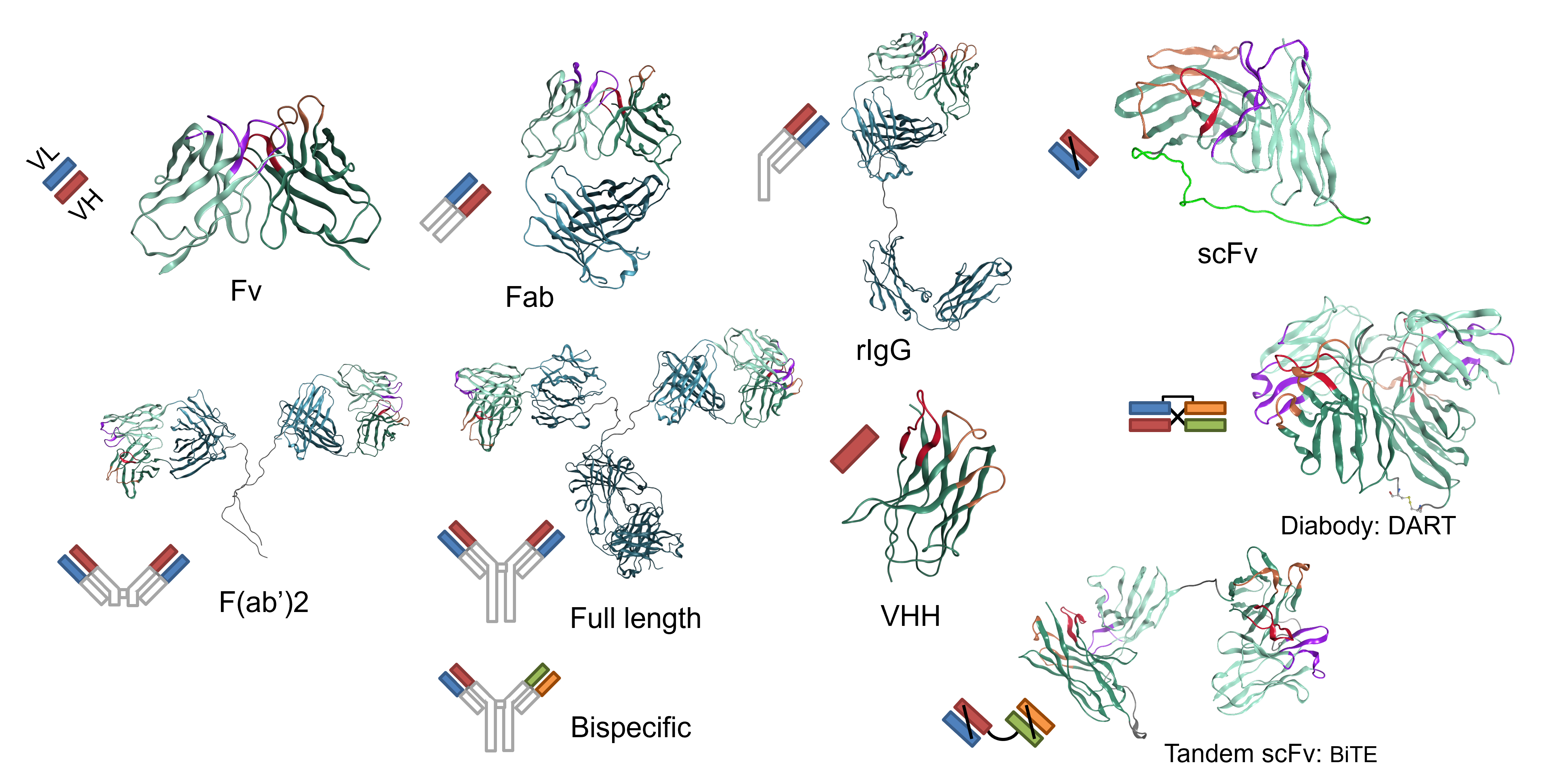
各種抗体のモデリング例
ループ/リンカーモデリング
ループ/リンカーモデリングは、タンパク質のループ構造を対話的に構築する機能です。ループ構造の構築手法として、PDBデータ内から検索する経験的探索と、新規のループ構造を構築するde novo探索の二種類が提供されています。溶媒和を考慮した相互作用エネルギーや、標的分子との親和性、電子密度マップとの適合性を加味して評価できます。ループモデリングは、CDRループのサンプリングに利用できます。リンカーモデリングでは、2つのタンパク質を融合するためのリンカー候補を探索します。この機能は、scFvのような融合タンパク質の構築に活用できます。
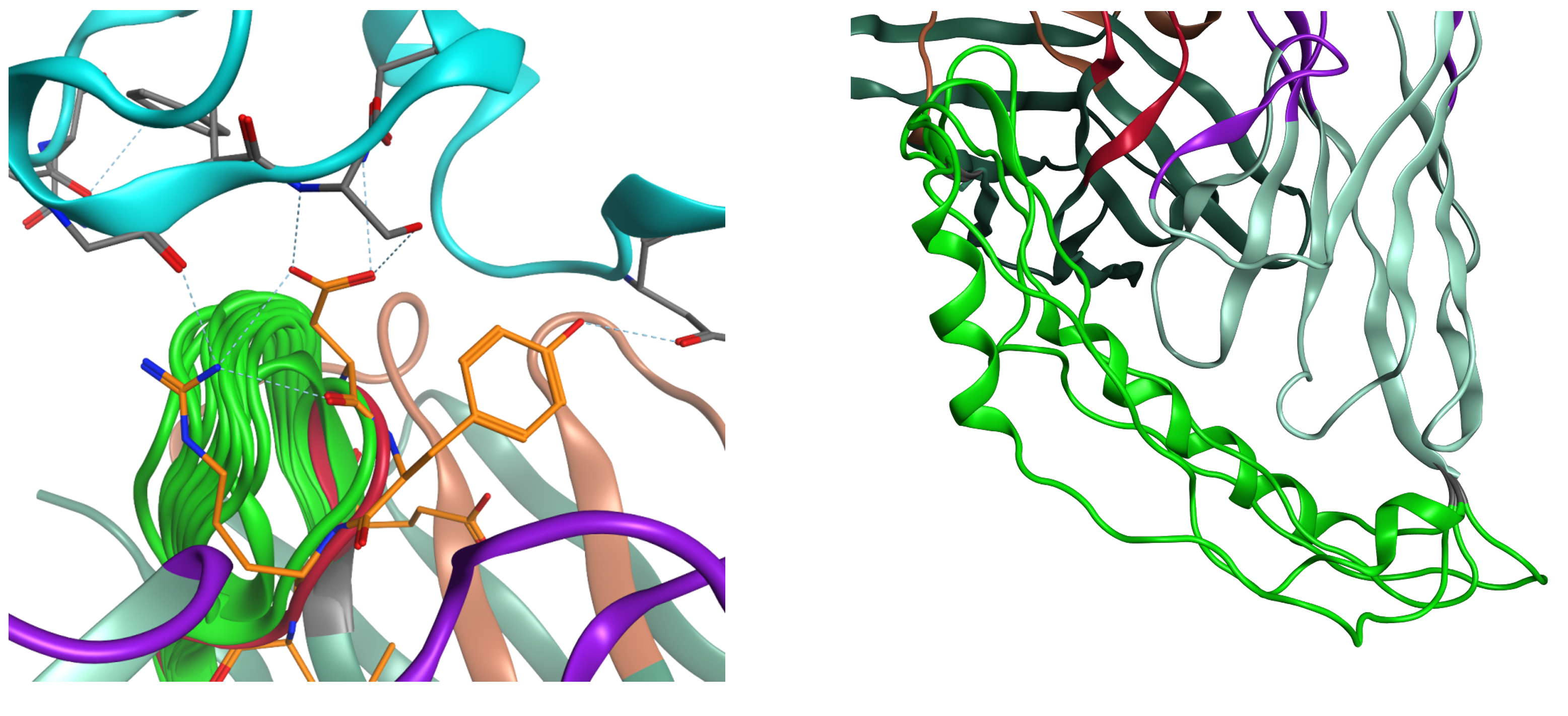
(左)ループモデリングによるCDRループ(緑)の検討(右)リンカーモデリングによるscFvのリンカー(緑)の検討
抗体データベース
MOE Project データベースの一つであり、最新版では約16,000のPDB構造が収録されています。各構造にはCDRやFRなどのアノテーションが付与されており、重要な領域が一目で確認できます。抗体の配列アラインメントとFR領域での立体構造の重ね合わせが行われており、比較解析がしやすくなっています。専用パネルを使用することで、キーワード、種、抗体や抗原の配列、立体構造、アノテーション情報などを基に構造を検索できます。種やCDRの残基長などの統計情報を確認できます。インハウスデータの追加も可能です。抗体データベースは抗体モデリングのテンプレートとして利用されます。
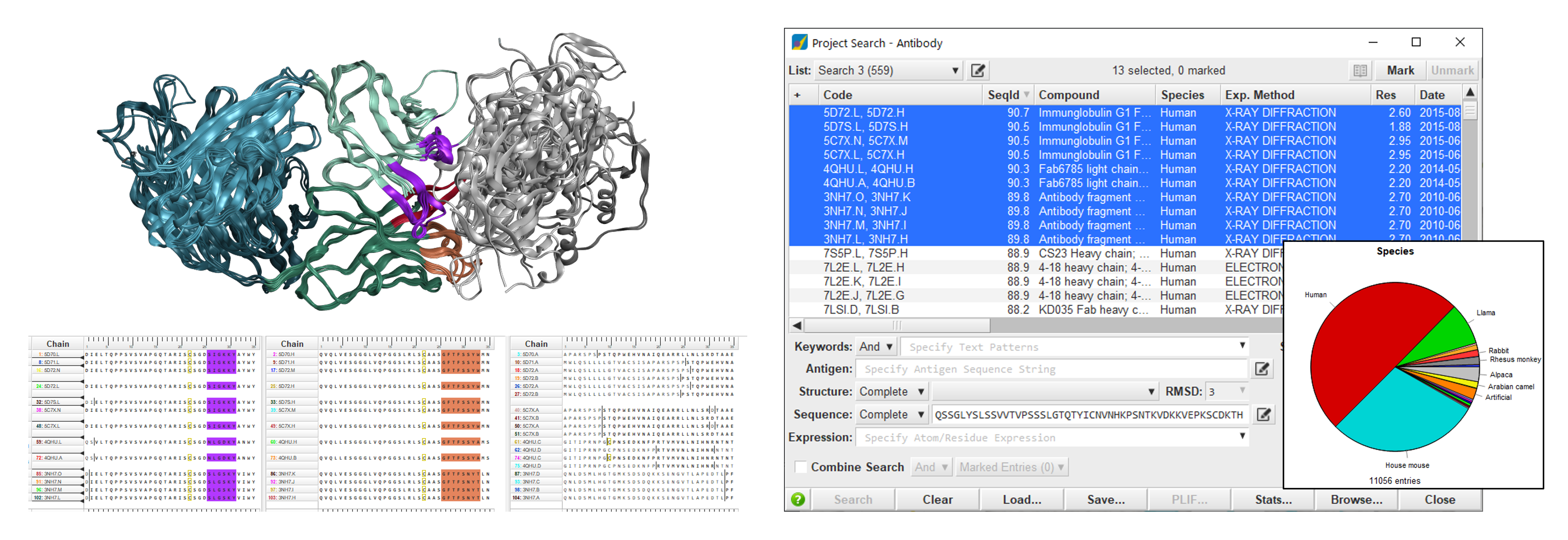
(左)アノテーション付けと重ね合わせがされた抗体構造(右)データベースの検索パネル
title:{タンパク質デザイン}
タンパク質変異体モデリング
立体構造を目視で確認しながら、ターゲットとなるアミノ酸残基を指定するだけで、変異体の配座候補を得ることができます。事前に用意された側鎖配座データベースから、立体障害のない候補構造を提案します。それぞれの候補は、ファンデルワールスエネルギーや水素結合エネルギーなどによってランク付けされます。非天然アミノ酸への変異も可能です。ユーザーオリジナルの非天然アミノ酸を登録し、変異体構築に用いることができます。この機能は抗体薬物複合体(ADC)のモデリングにも応用できます。
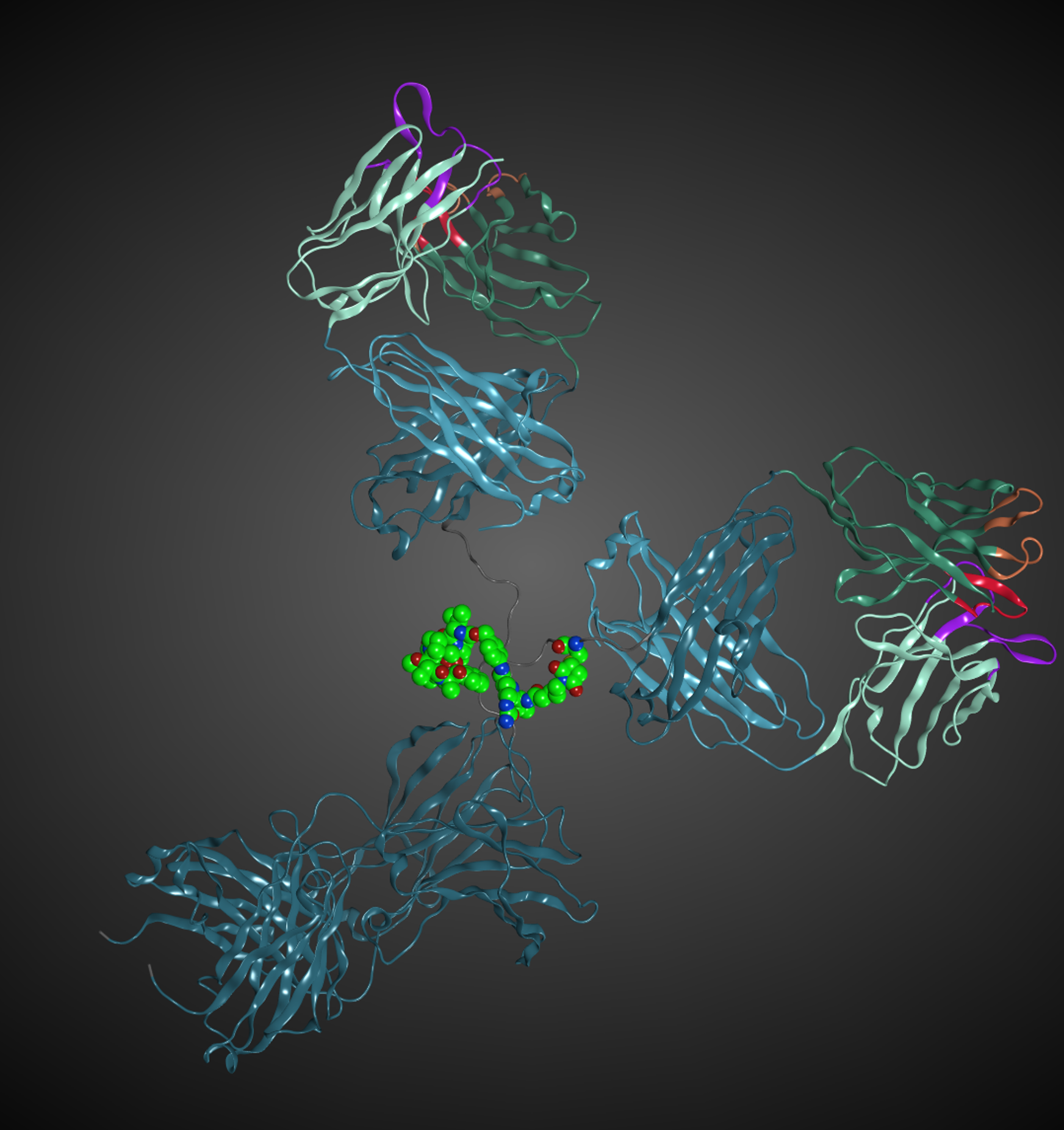
ADCのモデリング例。緑が低分子化合物(ペイロード)+リンカー+システインを非天然アミノ酸として導入した部分。
タンパク質デザイン計算
複数のタンパク質変異体モデルを一回の計算で構築して解析できます。リガンドとの親和性に重要な残基の解析(ホットスポット解析)や、タンパク質の熱安定性に影響を与える変異体の予測などが行えます。変異体の様々な物性値を推算し、野生型の物性値と比較することで、実験の目的に応じた変異体作製に貢献できます。右図は抗体のCDRのセリンを別のアミノ酸残基に変異し抗原との相互作用エネルギーが改善された例です(PDB ID: 1VFB)。グルタミン酸への変異により、抗原のリジンとの水素結合が新たに形成されています。また、タンパク質の熱安定性も同時に評価できます。

変異体解析の例
バーチャルファージディスプレイ
バーチャルファージディスプレイにより、リガンドとの親和性や熱安定性の向上、目的の物性値を持つタンパク質候補を効率的に構築することができます。ランダムサンプリングにより構築した変異体構造において各種スコアや物性値を計算し、それぞれの残基位置ごとの活性体に含まれるアミノ酸出現頻度と情報エントロピーを算出します。その結果をもとに、膨大な組み合わせの中から目的の物性にフォーカスしたライブラリーに絞り、変異体モデル構築します。
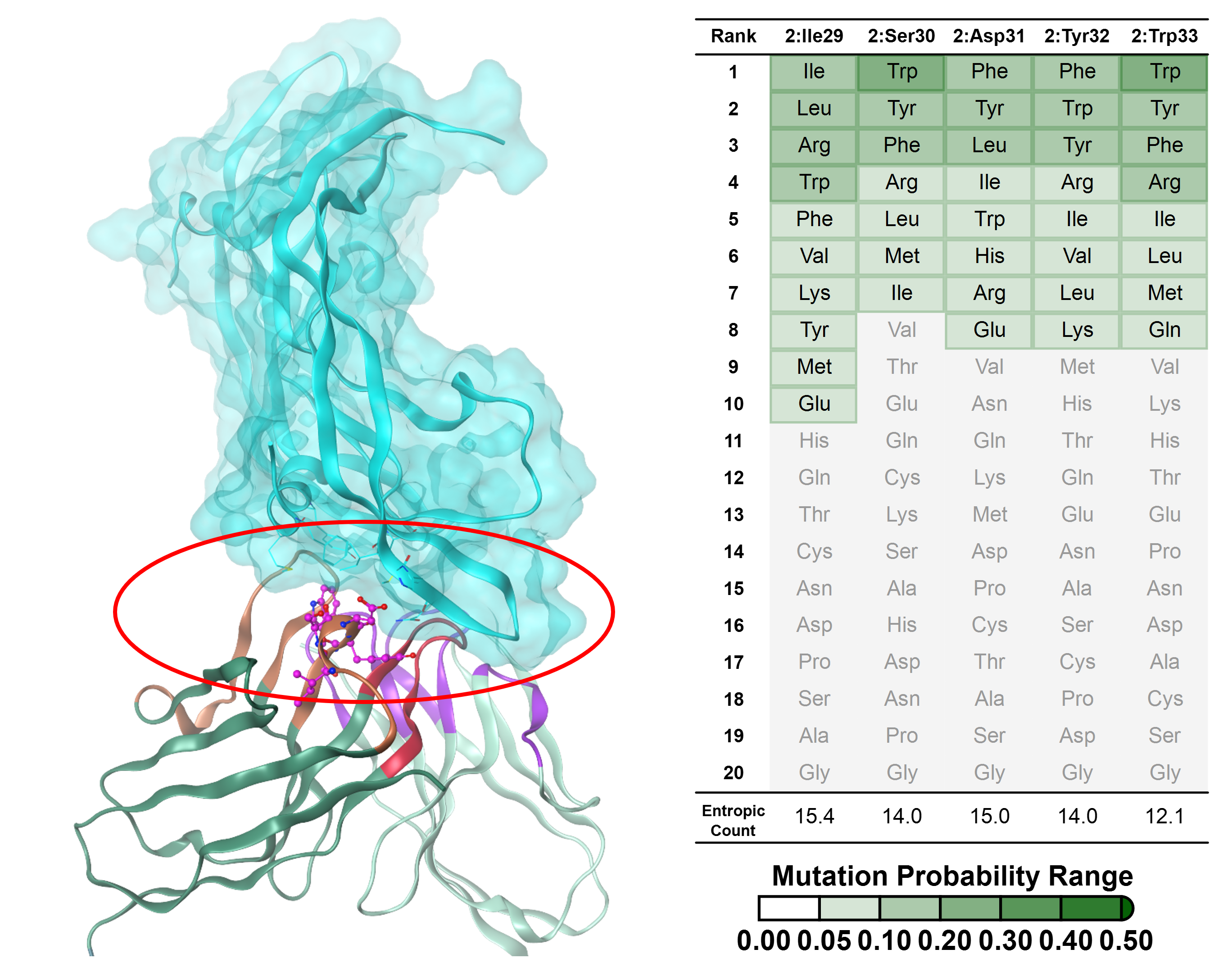
VEGF-G6 抗体(PDB ID: 2FJG)のCDR H1中の5残基について検討した例。表の一番上の行が野生型の残基。高ランクな残基ほど出現頻度が高い。
title:{タンパク質間相互作用解析}
網羅的な相互作用の検出
タンパク質の相互作用解析機能では、水素結合、イオン相互作用、ファンデルワールス相互作用、共有結合、CH…O相互作用、π…H相互作用、ハロゲン結合、配位結合、アレーン接触を検出できます。分子内相互作用やタンパク質間相互作用(PPI)を解析できます。残基番号を軸の値に利用した2次元グラフを表示できます。データ点を選択すると、対応する3次元構造を選択できます。

タンパク質間相互作用解析の例
タンパク質表面パッチ解析
疎水性、正電荷、負電荷の傾向が強いタンパク質表面の一部(表面パッチ)を可視化します。表面パッチは、タンパク質の凝集やタンパク質間相互作用、溶解度などに影響します。凝集を防ぎ溶解度を向上するための残基の特定や、PPI阻害剤のターゲットの探索に活用できます。表面パッチの二次元への投影図は、野生型と変異体との比較などに利用できます。
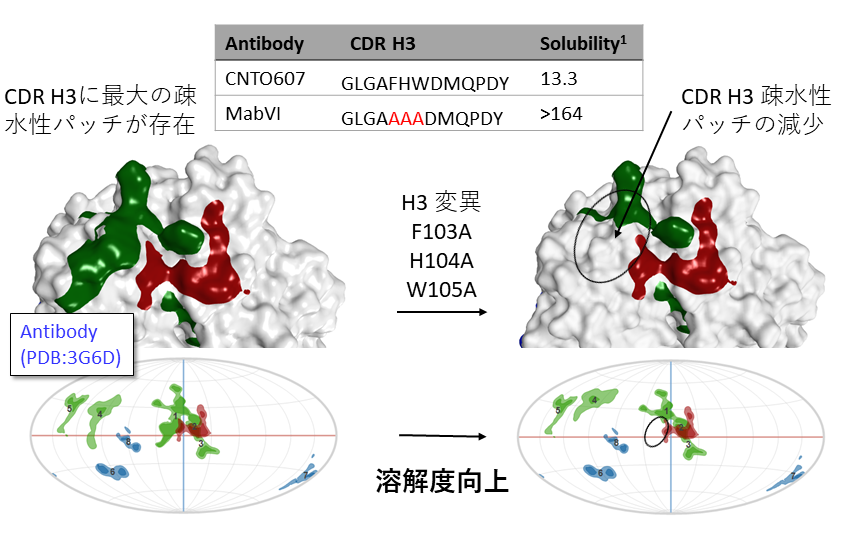
抗体CNTO607(左図)は高濃度で凝集。CDR H3にある疎水性パッチが凝集体形成の原因となりうる。疎水性パッチ領域にアラニンを導入した変異体(右図)は疎水性パッチが減少し溶解度の向上が見られた。 1. Wu, S.-J.; et al. Protein. Eng. Des. Sel. 2010, 23 (8), 643–651.
タンパク質-タンパク質ドッキング
タンパク質-タンパク質間のドッキングシミュレーションは、単体のタンパク質の構造から、それらが形成する複合体の構造を予測します。得られた複合体の構造はタンパク質の重要部位の特定(エピトープマッピング)、タンパク質間の相互作用の解析・設計に利用できます。計算は粗視化モデルとグリッドを用いて高速化しています。探索範囲を抗体のCDRや酵素活性部位などに限定して計算を行うことができます。
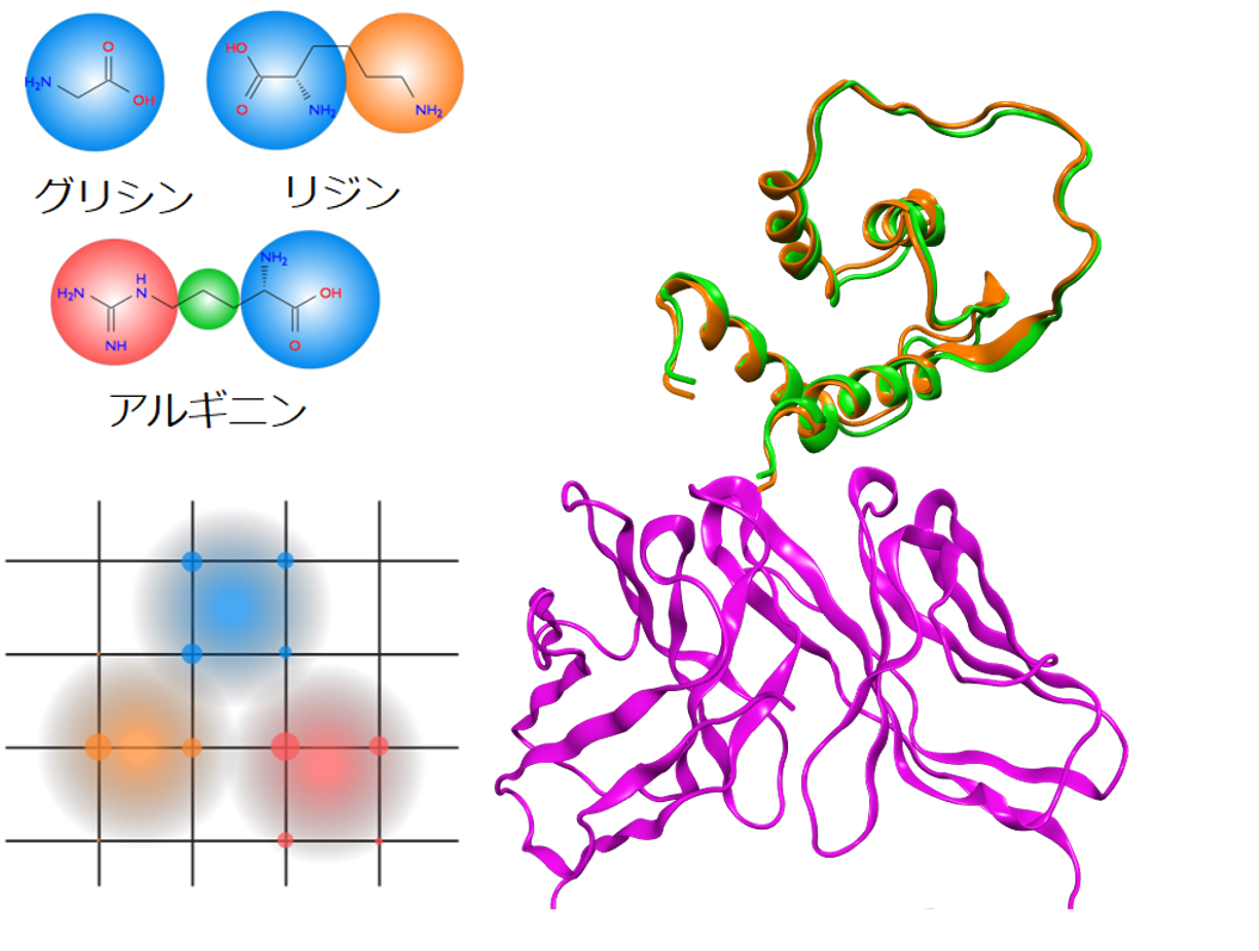
アミノ酸残基の粗視化イメージ(左上)、リガンドのガウス関数で重み付けされた密度のイメージ(左下)、抗体(右下、紫)とインターロイキン13(右上、実験結果:橙、計算結果:緑)のドッキング構造(PDB ID: 3G6D、RMSD: 0.8 Å)
エピトープマッピング
エピトープマッピングは抗原の抗体結合部位(エピトープ)を同定することです。同定されたエピトープの情報は、新たな抗体医薬を設計する上で大変有用です。MOEのエピトープマッピングの機能では、タンパク質-タンパク質ドッキングの結果に対して、表面パッチを取り入れた相互作用フィンガープリントを用いたクラスタリングと相互作用エネルギーによるランク付けを行って、エピトープのアミノ酸残基を予測します。
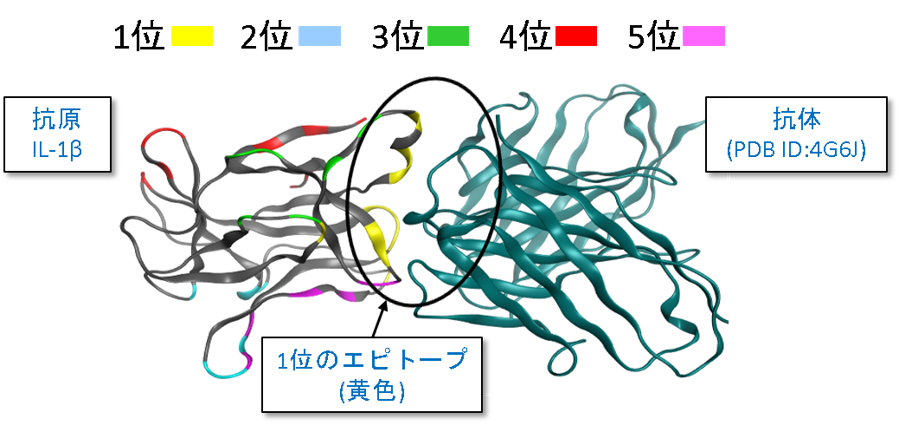
インターロイキン-1β(IL-1β)の上位5件のエピトープ予測例
ファーマコフォアによるバーチャルスクリーニング
生物学的製剤と標的分子との重要な相互作用を特定し、それらをファーマコフォアモデルに変換できます。その際、標的分子の形状を考慮できます。数千あるいは数百万のタンパク質構造に対してファーマコフォア検索(マッチング)を行い、標的分子と結合し得る生物学的製剤を見出すことができます。CDRなどタンパク質の一部のみを検索対象とすることで、高速かつ効率的に探索できます。この機能は、タンパク質間ドッキングの高速な代替法として利用できます。
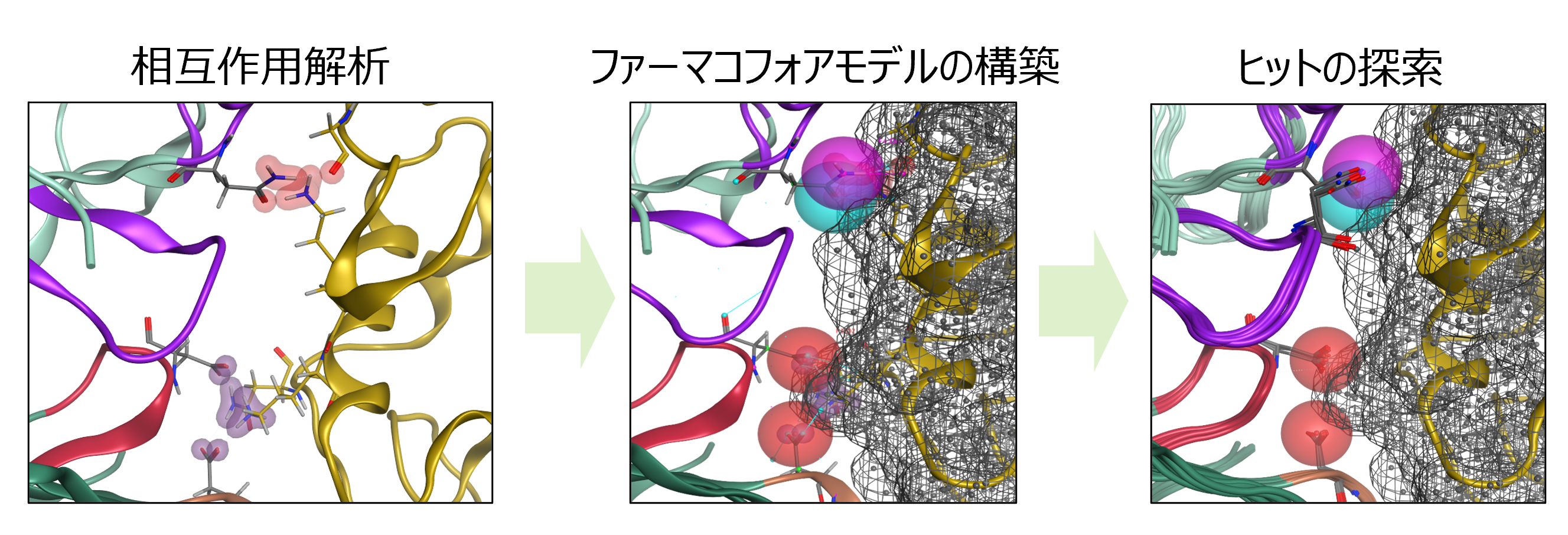
ファーマコフォアによるバーチャルスクリーニングの流れ
title:{Developabilityの評価}
タンパク質物性推算/タンパク質記述子
112種類のタンパク質用の記述子が計算できます。例えば、二次構造の含有率、疎水性・親水性表面積、ファンデルワールス体積、等電点、総電荷、ゼータ電位、疎水性/正電荷/負電荷表面パッチの面積などを計算できます。抗体においては、表面パッチの面積などをCDRに限定した記述子が使用できます。生物学的製剤の品質に関連するものとして、主鎖アミドのpKa、メチオニン残基周辺の水分子の数、Cα/側鎖のRMSDなどを計算できます。これらは記述子として、タンパク質の物性予測モデルやdevelopabilityの評価に利用できます。物性値の計算は、グランドカノニカルアンサンブルによるサンプリングに対応しており、pHや構造変化を考慮した物性推算が可能です。
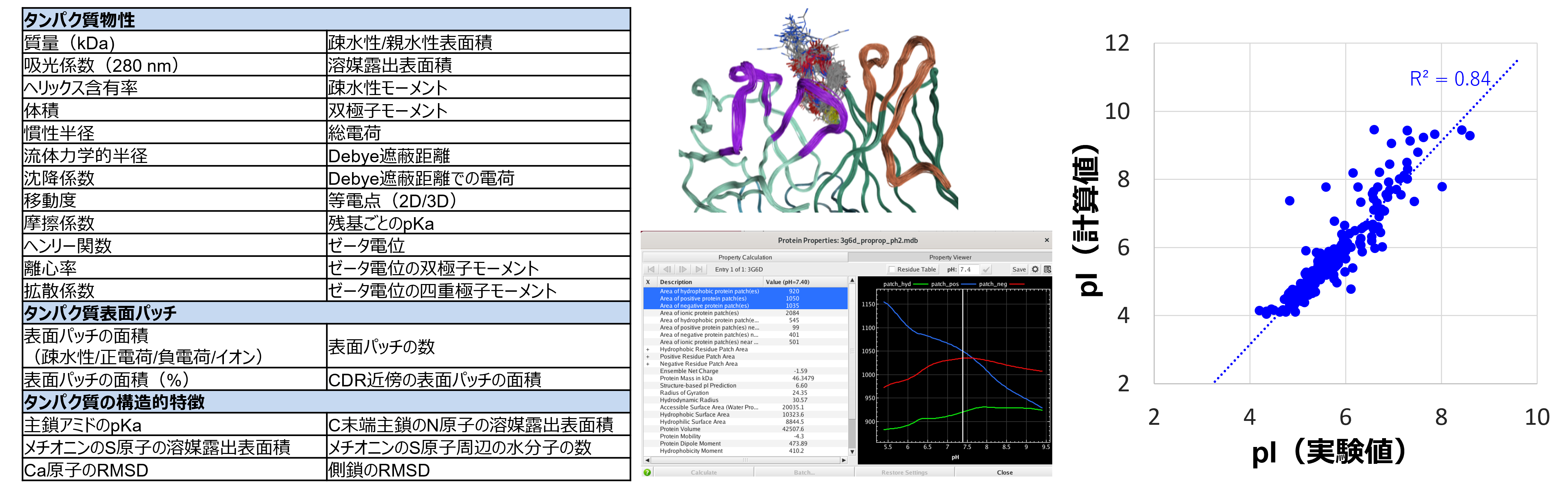
(左)タンパク質記述子の例。(中)pHと構造変化を考慮した物性推算の例。(右)235個の様々なタンパク質構造における等電点(pI)の実験値と予測値の相関図。
物性予測モデルの構築
計算したタンパク質記述子を基に、物性を予測する統計モデルを構築できます。溶解度などの物性値を予測する線形モデル、分解される/されないといった二値データを予測する確率モデル、記述子のとる値が閾値を超える/超えないで分類を行う決定木モデルを構築できます。
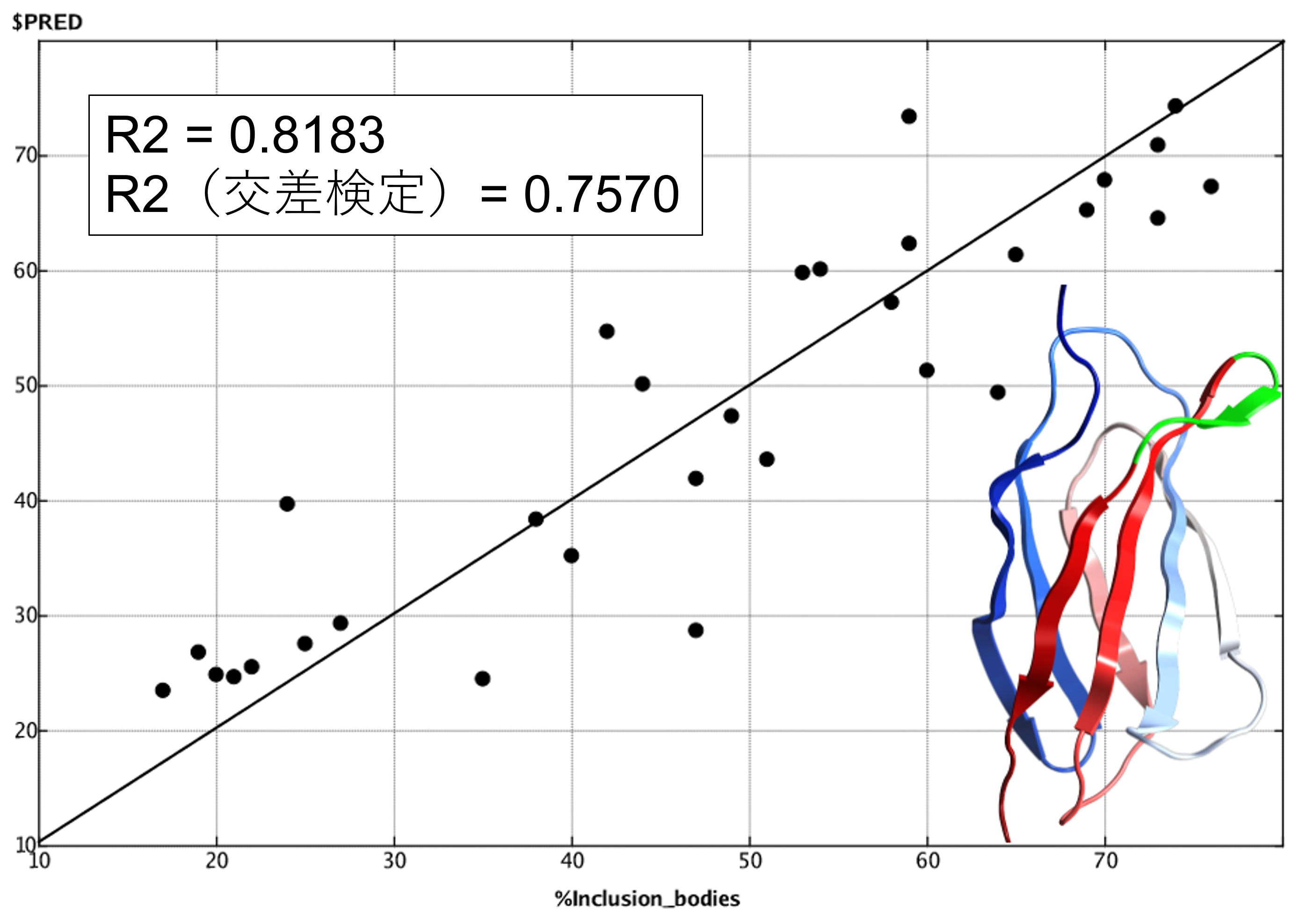
FGループの配列が異なる31個のアドネクチンの溶解度の予測モデル。縦軸が予測値、横軸が実験値。予測モデルには、疎水性モーメント、疎水性表面パッチ、正電荷表面パッチに関する4個のタンパク質記述子を使用。
}:tab


