MOE - ケモインフォマティクスとQSAR
tab:{
title:{概要}
操作性に優れた分子データベース機能を利用して、大規模な分子ライブラリーを取り扱うことが可能です。脱塩、立体構造の立ち上げといった前処理や、物性値、フィンガープリントの算出など一括で処理できます。クラスタリングやQSAR、合成ルールによるコンビケムライブラリーの構築など、ライブラリー設計からリード化合物の探索、リード最適化に至るまでの創薬研究において利用される多くの機能を提供しています。
title:{分子データの標準化}
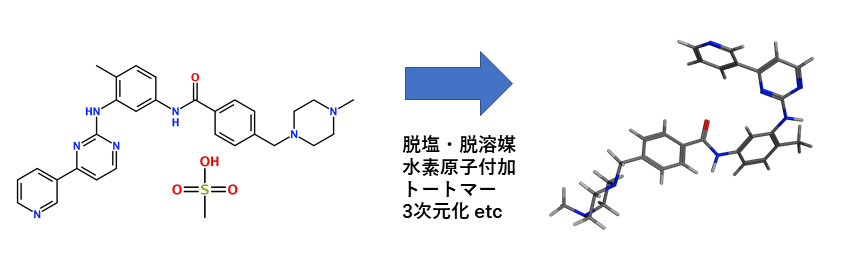
多数の分子構造データを統一的な条件下で取り扱うための標準化機能を搭載しています。多数の分子構造を含むライブラリーに対しても高速に一括処理を行い、素早く解析プロセスに移行できます。また、コマンドラインから利用できるツールもあわせて搭載しており、ライブラリーの管理システムにも容易に組み込めます。
分子構造の標準化
分子構造データに含まれる塩・溶媒分子の削除や、pHを指定した水素原子の付加、中性化、結合長の調整といった分子構造の標準化処理が簡単に行えます。3次元構造から適切な2次元構造を抽出することも可能です。
分子構造の3次元化
データベース中の分子構造にエネルギー極小化計算を行います。2次元構造からの立体構造立ち上げ時には、独自の4次元Minimizerを用いることで衝突による異常構造を避けた効率のよい3次元構造への変換を行うことができます。
SDコマンドラインツール
SDファイル(*.sdf)形式に登録された分子構造データに対して各種標準化を行うコマンドを利用できます。大量データのバッチ処理や、管理システムに組み込んだ自動的な標準化処理も容易です。
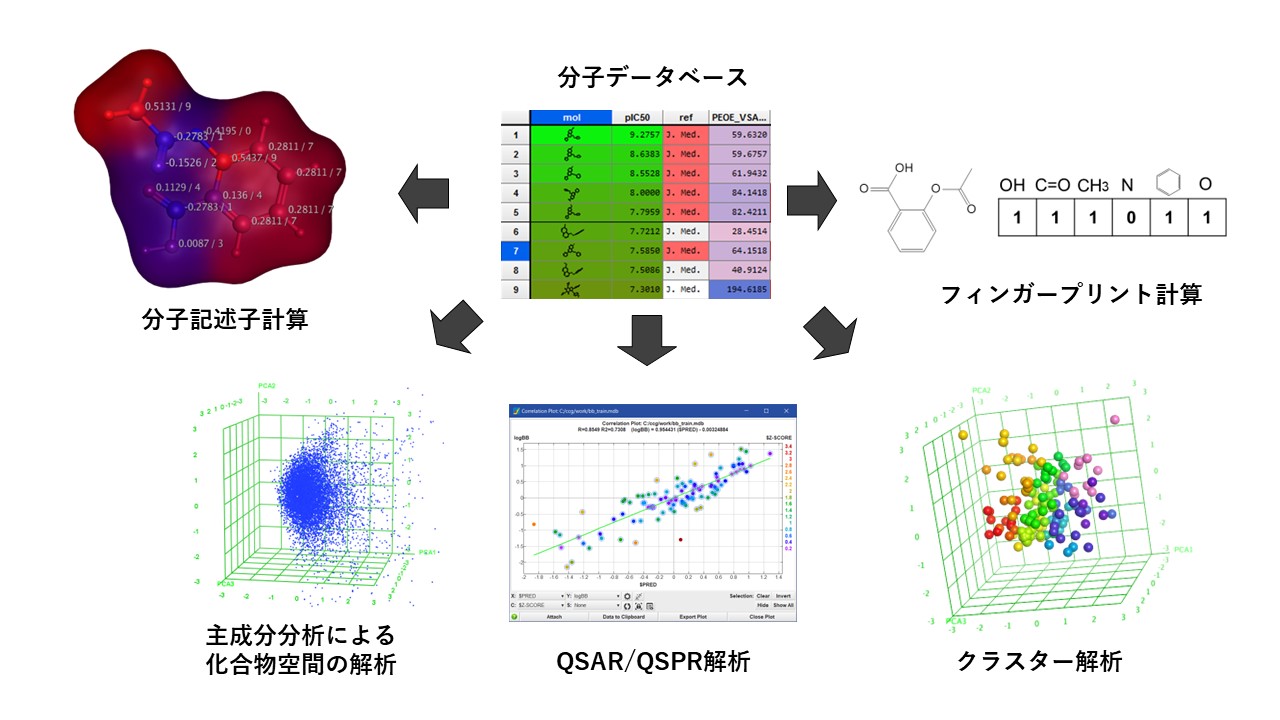
title:{分子記述子計算}
MOEの分子データベースに保存された分子構造に対して、様々な記述子を計算し、QSARモデルの構築やライブラリーの解析に利用することができます。記述子には、物性、原子数、結合数、電荷、エネルギー、表面、形状、グリッド、ドラッグライク、コネクティビティ、インデックス、マトリックス、ファーマコフォア、VolSurf、MOPAC等の2D/3D構造情報を用いる400種類以上が標準で用意されています。タンパク質解析用の記述子も用意されています。ユーザー定義のカスタム記述子を作成できます。
VSA(van der Waals surface area)記述子
MOE独自の記述子で、logP、モル屈折、電荷の特性に関し、ある一定の特性レンジに入るような分子の表面積を記述子の値として使用します。2次元情報だけから3次元的な特性を算出するので2.5D記述子と言われ、ライブラリーの解析、QSAR/QSPRモデルの構築まで利用できる汎用性の高い記述子です。
統計解析プロット
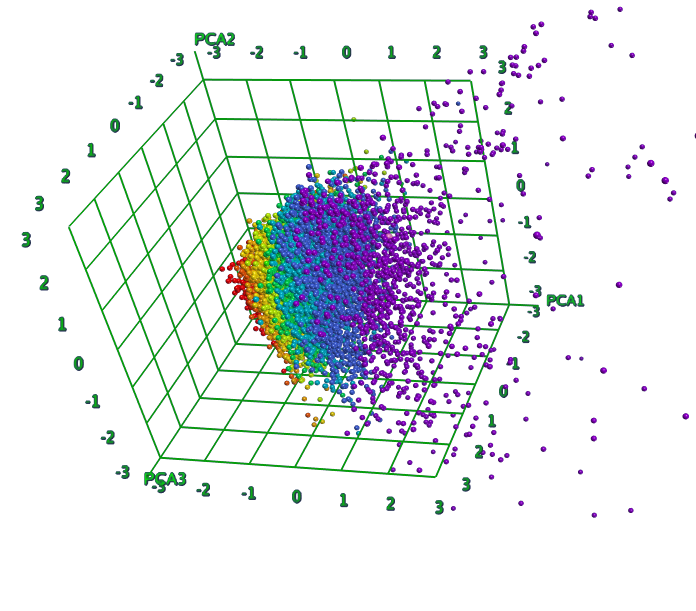
2D相関図、ヒストグラム、相関マトリックスの表示、主成分分析の各成分による3Dプロット、各記述子の相対的な重要性を比較し出力する機能等、算出した記述子を使用してデータベースの統計的な解析と表示を行えます。
title:{QSAR/QSPRモデルとベイジアン分類モデル}
化合物の構造から計算される記述子より、活性予測モデルあるいは物性予測モデルを構築できます。精度のある活性値から作成される線形モデルはリード最適化の段階で有効です。また、比較的粗いデータの処理に適した確率モデルや分類モデルはリード探索の段階で有効です。AutoQuaSAR (AutoQSAR)を用いることで、通常専門家が行う手順を自動化し、ユーザーは分子構造と実験値を入力するだけで適切なQSAR/QSPRモデルを構築できます。
線形モデル
回帰分析(PCR、PLS)を行い、記述子と活性値との相関式(線形モデル)を構築します。構築したモデルを使い新規化合物の活性値を予測することができます。Cross Validationによるモデルの検証も行えます。QuaSAR-Evolutionを用いることで、遺伝的アルゴリズムを用いて多数の予測モデルの中より記述子を選択し重回帰分析によりモデル化が可能です。
確率モデル(バイナリモデル)
従来の回帰分析ではモデル化の難しい比較的精度の低いデータ、例えばHTS実験で得られる活性の「有る」「無し」というようなバイナリデータ(2値データ)をモデル化する手法として、独自のBinary QSARを搭載しています。Binary QSARは、ベイズの定理をQSARに応用することにより、構造活性相関を確率モデルとして表します。Binary QSARは、実験誤差やアウトライヤーの存在による影響を受けにくく、大量のデータ処理を要求されるリード化合物のスクリーニングにおいて特にその効果を発揮します。
分類モデル
記述子のとる値が閾値を超えるか超えないかで分類を行う決定木を作成します。活性のタイプでライブラリーが分類されている場合や、HTS実験から活性の強度で幾つかのクラスに大まかに分類されている場合に、数種のクラスに分類するようなモデルを構築し、サンプルを分類するのに有効です。
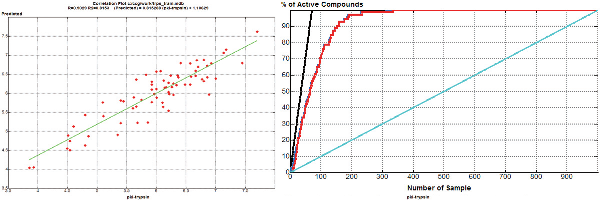
title:{化合物の類似性/ダイバーシティ/クラスタリング}
フィンガープリント計算
規定された特徴の有無によって化合物を表現するフィンガープリントを算出します。MACCSキーによる部分構造フィンガープリント、ファーマコフォアによるフィンガープリント、分子形状によるフィンガープリントの3タイプ11種類を利用できます。フィンガープリントを利用することで大量の化合物データベースに対する類似構造検索やクラスタリングも高速に計算されます。
ファーマコフォアフィンガープリント
ファーマコフォアのタイプと各ファーマコフォア間の距離で表現されるフィンガープリントで、分子のもつ特性の相対位置の情報を含みます。距離の定義は2次元結合数距離と3次元距離の2種類があります。2次元結合数距離を利用することで、3次元構造に依存しないライブラリーの解析が可能です。
クラスター解析
記述子や物性値、フィンガープリントをもとにクラスタリングを行うことができます。記述子や物性値に関してはセルベースのクラスタリングを行います。フィンガープリントに関してはJarvis-Patrick法による距離に基づくクラスタリングを行います。
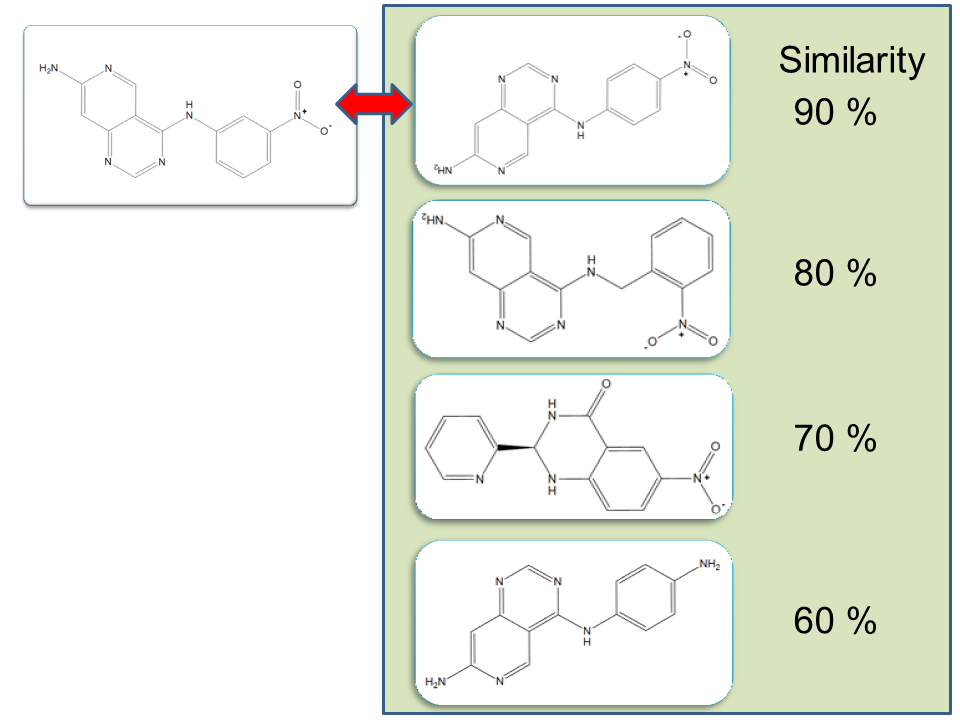
類似性・多様性解析
フィンガープリントの類似性を計算し、類似化合物をデータベースから検索します。また、フィンガープリントや記述子の値を使い化合物間の距離を計算し多様性のある部分集合を抽出します。ライブラリーをクラスターに分けている場合は、各クラスターから同数の部分集合を抽出することもできます。