MOE - 構造生物学
tab:{
title:{生体高分子の構造解析}
X線結晶解析やクライオ電子顕微鏡で得られた電子密度マップを読み込み、描画することができます。分子モデルを電子密度マップと比較しながら、マップとの適合性を確認できます。結晶セルにおける隣接原子を表示したり、水分子の位置を予測したりすることで、より詳細な構造解析を支援します。
電子密度マップの描画
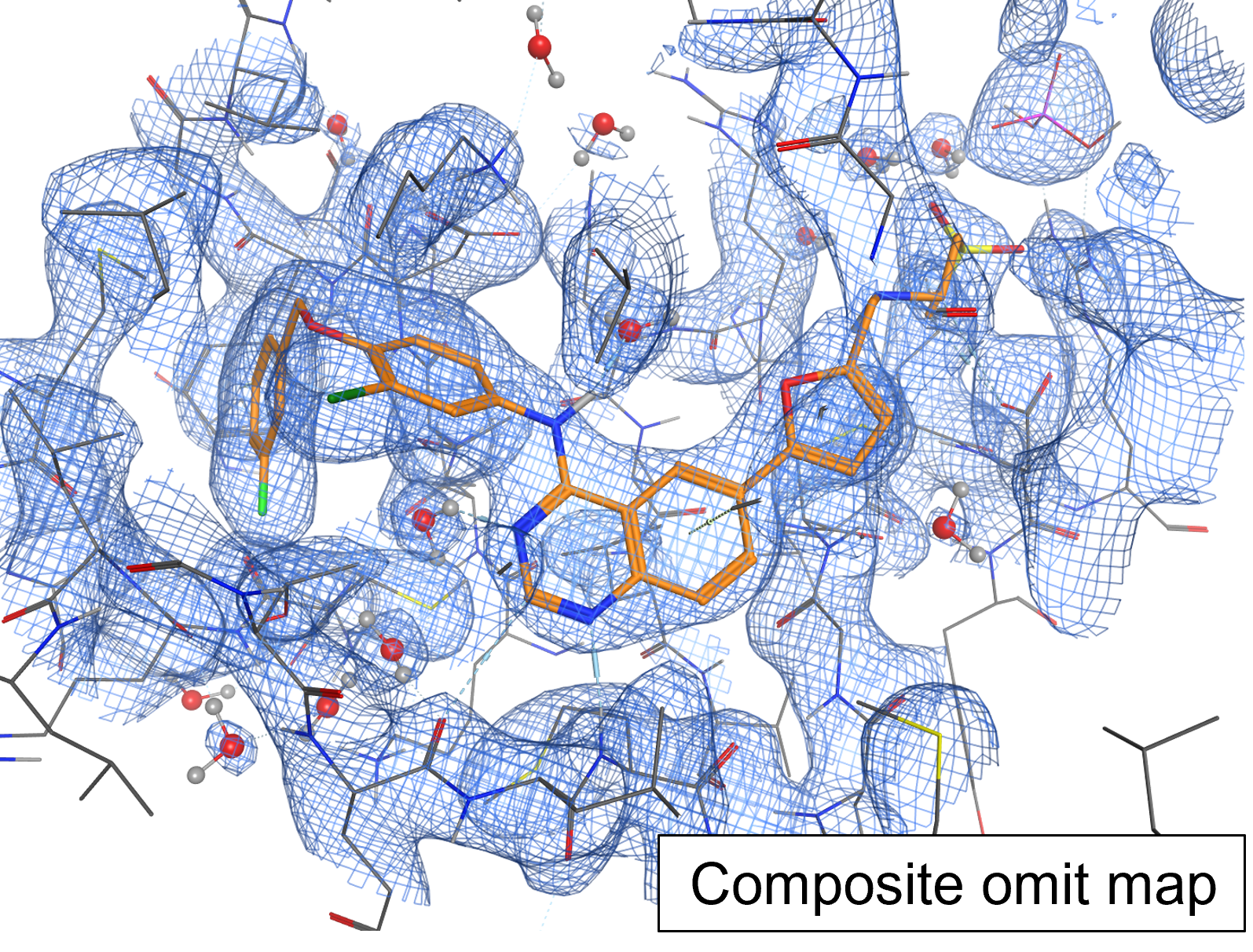
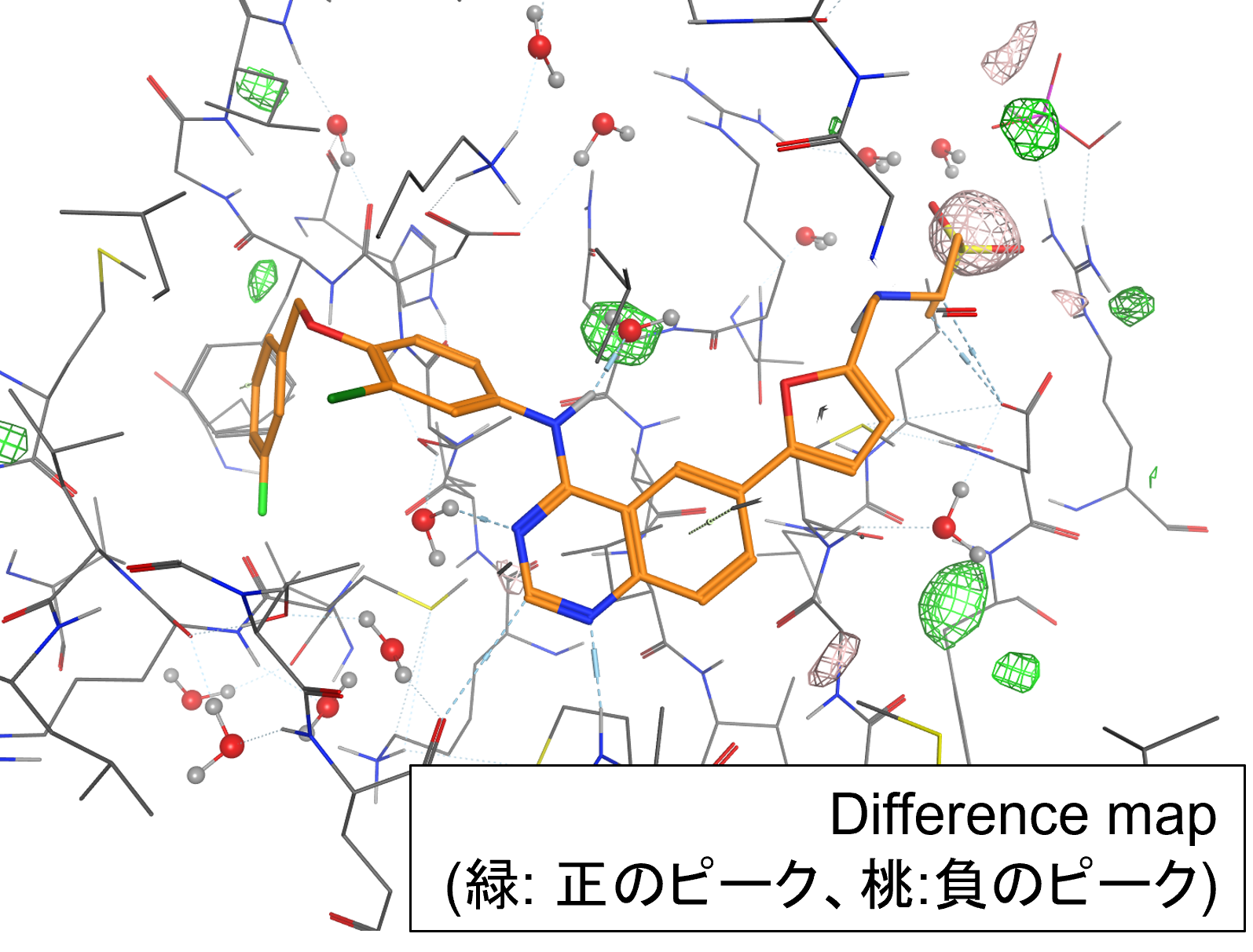
X線結晶解析やクライオ電子顕微鏡で得られた電子密度データを可視化します。X線結晶解析で得られた電子密度マップについては、composite omit map(2mFo-DFcマップ)やdifference map(mFo-DFcマップ)も表示することができます。電子密度マップと構造モデルを比較することで、原子が欠損または誤認識されている可能性のある領域を特定できます。
結晶格子と結晶構造における原子接触の可視化
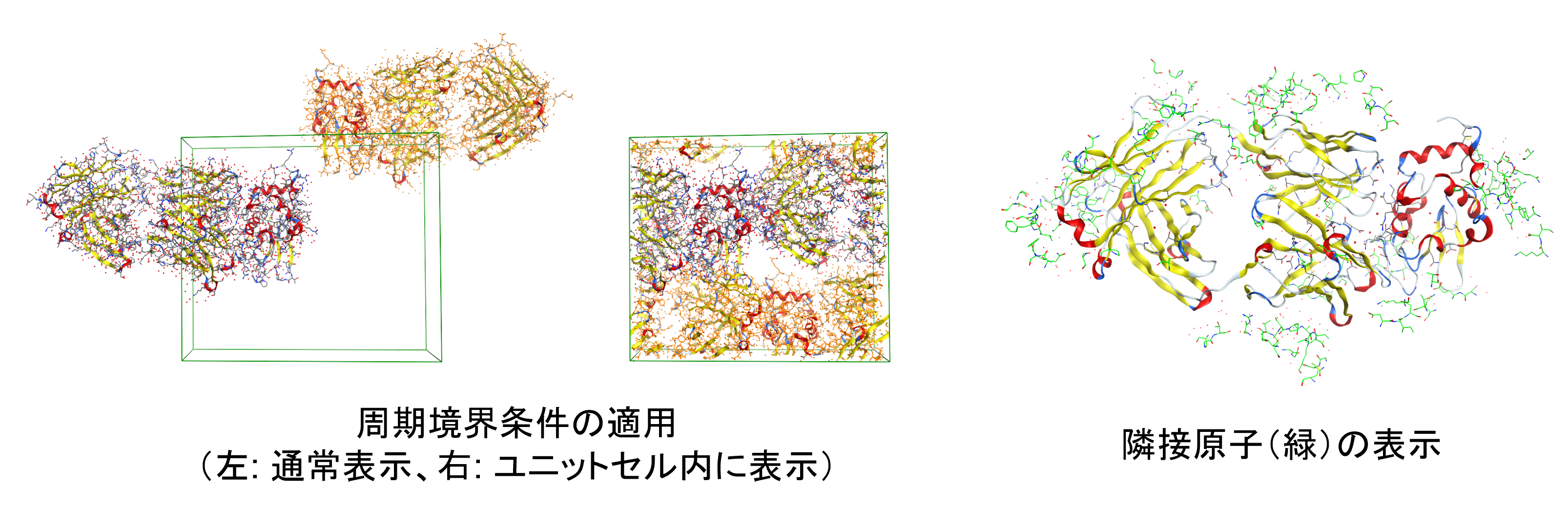
PDBデータに保存された結晶パラメータによる周期境界条件を適用したり、結晶セルにおける隣接原子を表示したりすることができます。結晶パッキングに伴う分子間相互作用の理解に役立ちます。
水分子の位置の予測
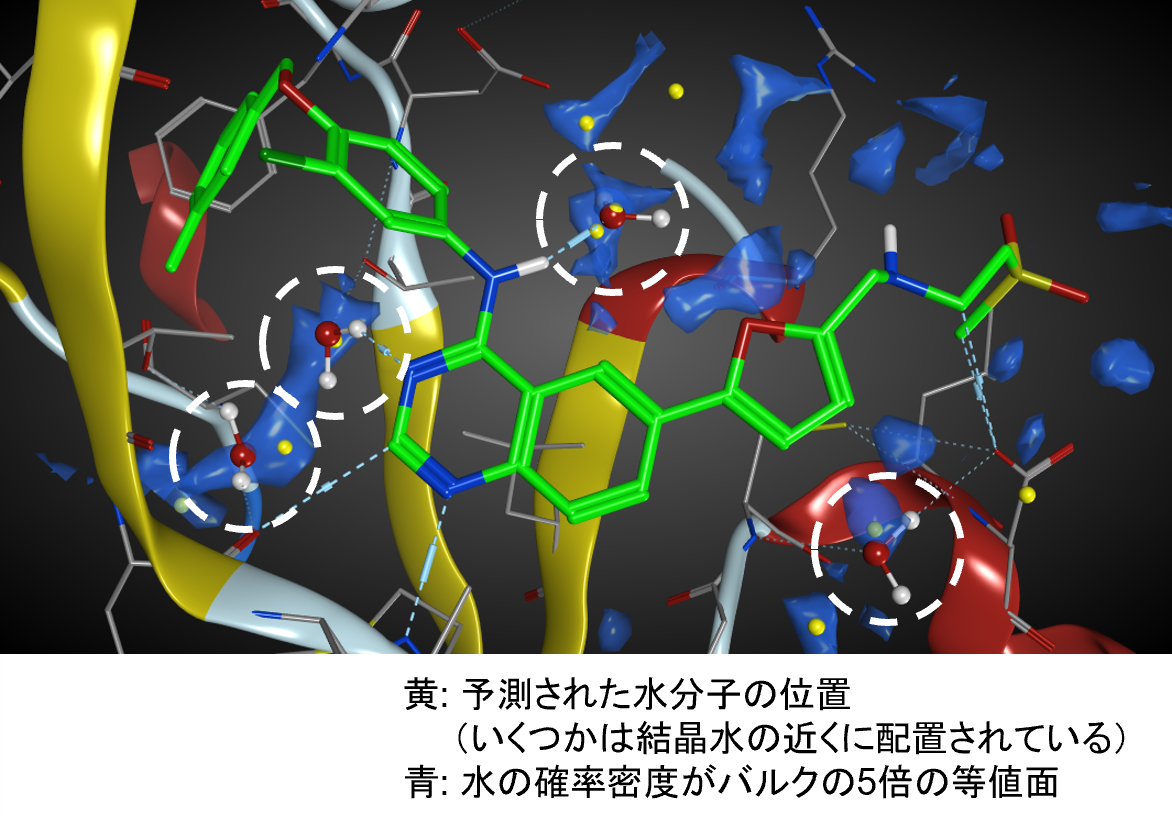
3D-RISM法により、高速かつ高精度に水分子の密度分布や溶媒和自由エネルギーを求め、タンパク質やリガンドの周辺に存在する水分子の位置を予測し、可視化します。水素結合の評価、水による相互作用ブリッジの理解、疎水性相互作用など、相互作用に関わる溶媒効果も詳細に検討できます。
MOE Solvent Analysis
title:{タンパク質・複合体構造のモデリング}
X線結晶構造解析における分子置換法での分子モデルをMOEのホモロジーモデリングにより構築できます。また、低解像度領域のタンパク質の柔軟なループ構造やリガンドの結合ポーズを電子密度マップと適合するようにモデリングを行い、誤りのない分子モデルを構築することもできます。
分子置換のためのホモロジーモデリング
MOEのホモロジーモデリングは、構造既知である相同タンパク質をテンプレートにして、構造未知のタンパク質立体構造を予測します。予測した構造をX線結晶構造解析における分子置換法での分子モデルとして利用できます。
ループ/リンカーモデリング
MOEのループ/リンカーモデリングは、タンパク質のループ構造や、2つのタンパク質をつなぐリンカー構造を探索します。経験的探索法、またはde novo探索法により任意の残基長のループ/リンカー構造を構築します。構築した構造に対して、溶媒和を考慮した相互作用エネルギーによる評価ができる他、電子密度データを使用して電子密度マップとの適合性による評価もできます。
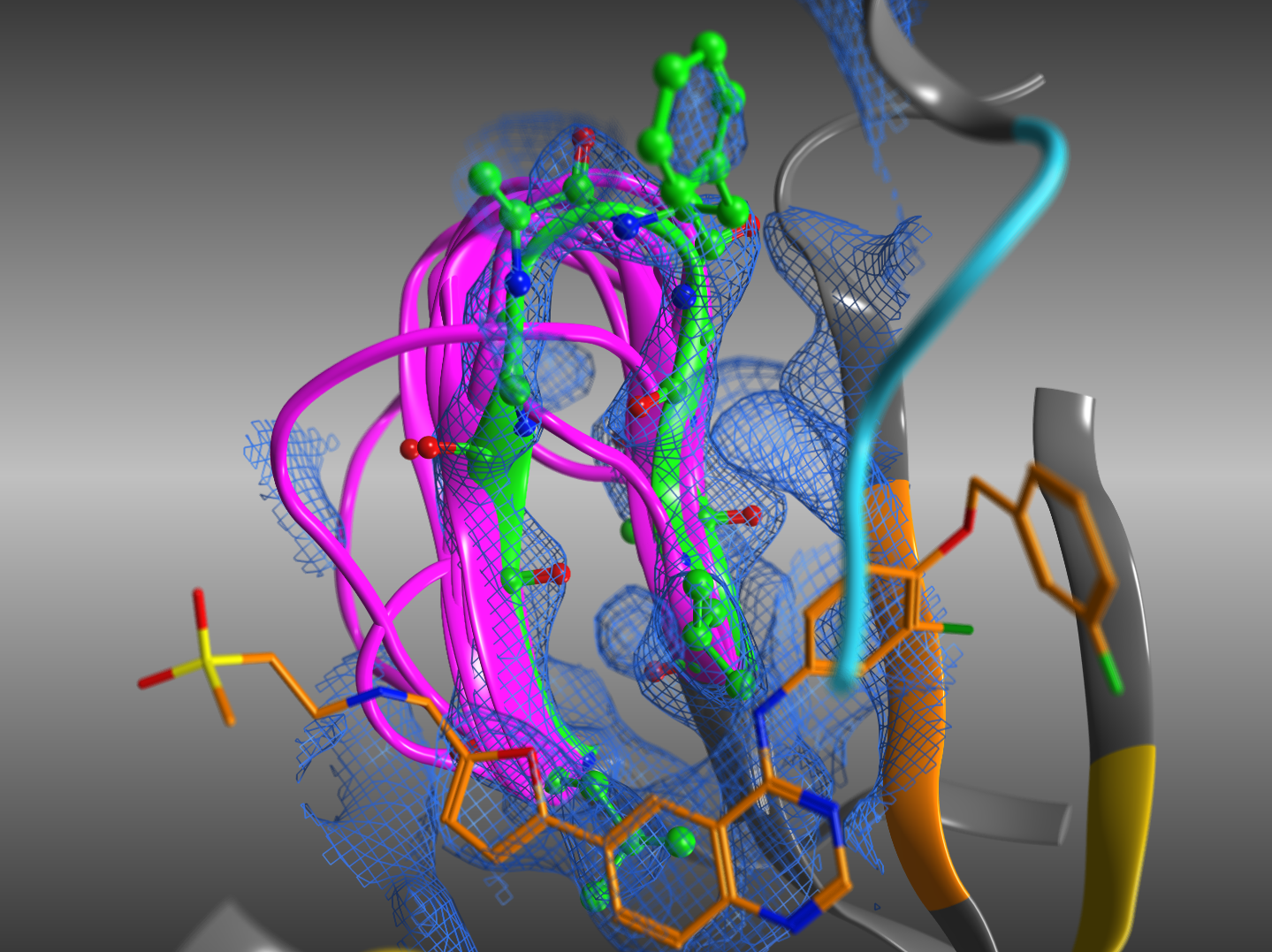
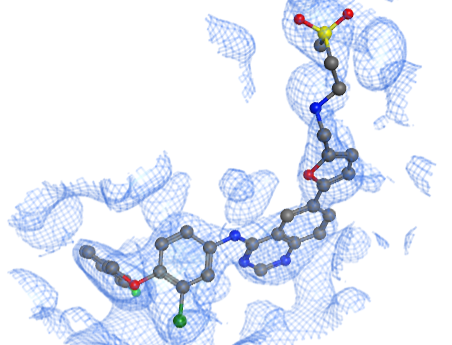
電子密度データを使用したドッキングシミュレーション
リガンドのドッキングシミュレーションにおいて電子密度データを使用できます。これにより、電子密度と重なるようにリガンドの配置と構造最適化計算が行われます。電子密度とのフィッティングを最大化しながら、歪みエネルギーを最小化したリガンドポーズが得られます。また、電子密度との重なりに基づくスコアリング関数も利用できます。
title:{タンパク質立体構造の評価・解析}
構造決定されたタンパク質立体構造、あるいはモデリングしたタンパク質立体構造のジオメトリの妥当性を評価したり、タンパク質の配列データをMOEのタンパク質ファミリーのアラインメントデータベースと比較して進化的関連を解析したりすることができます。
タンパク質立体構造の妥当性の評価
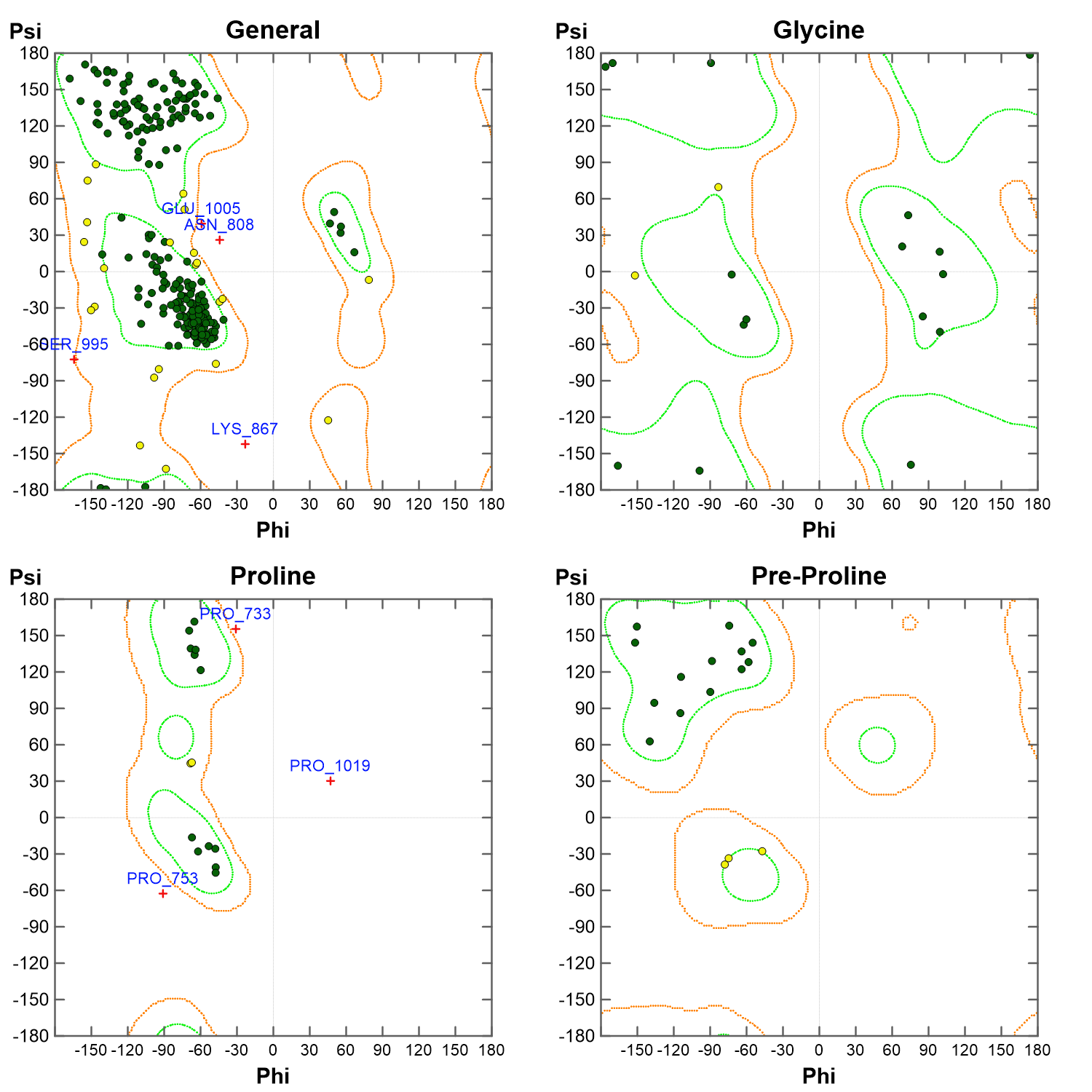
X線結晶解析データから統計的に導かれたデータを元に、モデリングされたタンパク質立体構造の妥当性を評価することができます。Ramachandranプロット(φ-ψプロット)では、残基毎の二面角の分布や、異常値を持つ残基をグラフィカルに分かりやすく表示します。また、二面角、結合距離、結合角などの立体化学的な情報をレポートとして出力し、統計的に異常な値を検出します。検出された情報を元に、異常構造を持つ部分にターゲットを絞って構造最適化計算を行うことにより、タンパク質立体構造の修正を簡単に行うことができます。
タンパク質ファミリーのアラインメントデータベース
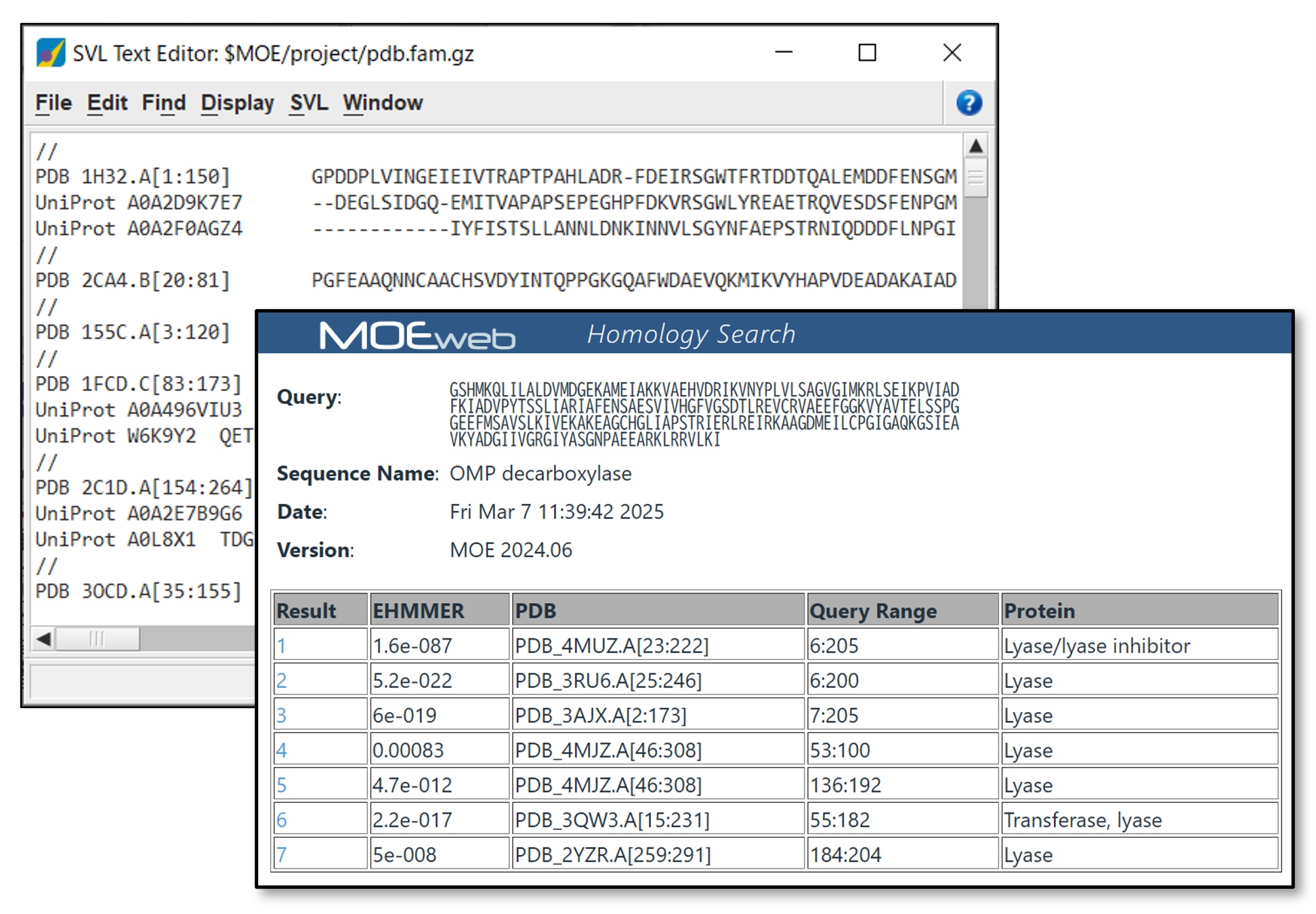
ホモロジーモデリングのために構築されたタンパク質ドメイン構造データベースとUniProtの配列データを用いて、配列および立体構造ベースのクラスタリングにより作成された、タンパク質ファミリーの配列データベースです。このデータベースを使用して遠縁の構造類似タンパク質の検出やファミリー単位の解析を行うことができます。ウェブブラウザから検索を実行できるMOE/webアプリケーションも提供されています。
}:tab


