PSILO
(タンパク質立体構造情報データベースシステム)
tab:{
title:{概要}
PSILO(Protein SILO)とは?
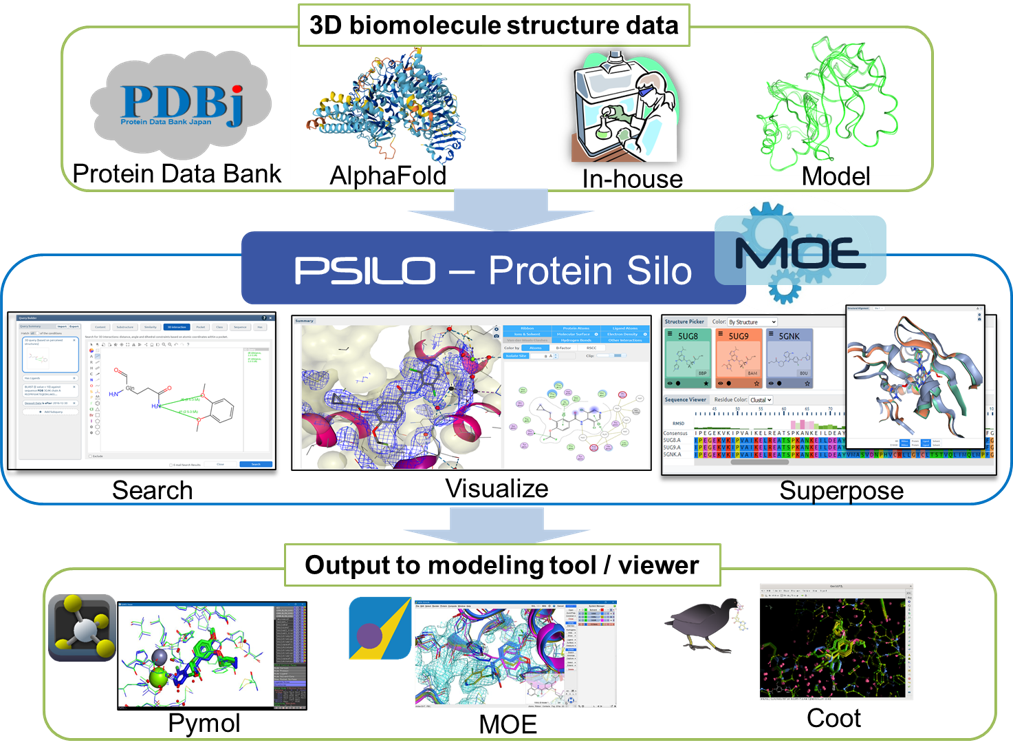
PSILOは、タンパク質、核酸、タンパク質-リガンド複合体などの生体高分子の立体構造情報を管理するデータベースシステムです。分散する公共データやインハウスデータなどを統合・整理して、多様な条件で検索可能にすることで、研究者間の情報共有を支援します。ユーザーは、データの検索、閲覧、登録、管理などの操作をウェブブラウザから簡単に行えます。
パンフレット
title:{機能}
検索
高速なテキスト検索
単語のインデクシングにより、googleライクな高速なテキスト検索を行います。
タンパク質/抗体アミノ酸配列検索
BLASTを内蔵しており、タンパク質、抗体の重鎖/軽鎖にターゲットを絞った配列検索を行います。
類似ポケット構造検索
配列やフォールディングに依らず、Cαの位置の重ね合わせから、類似の活性部位を持つタンパク質を検索します。
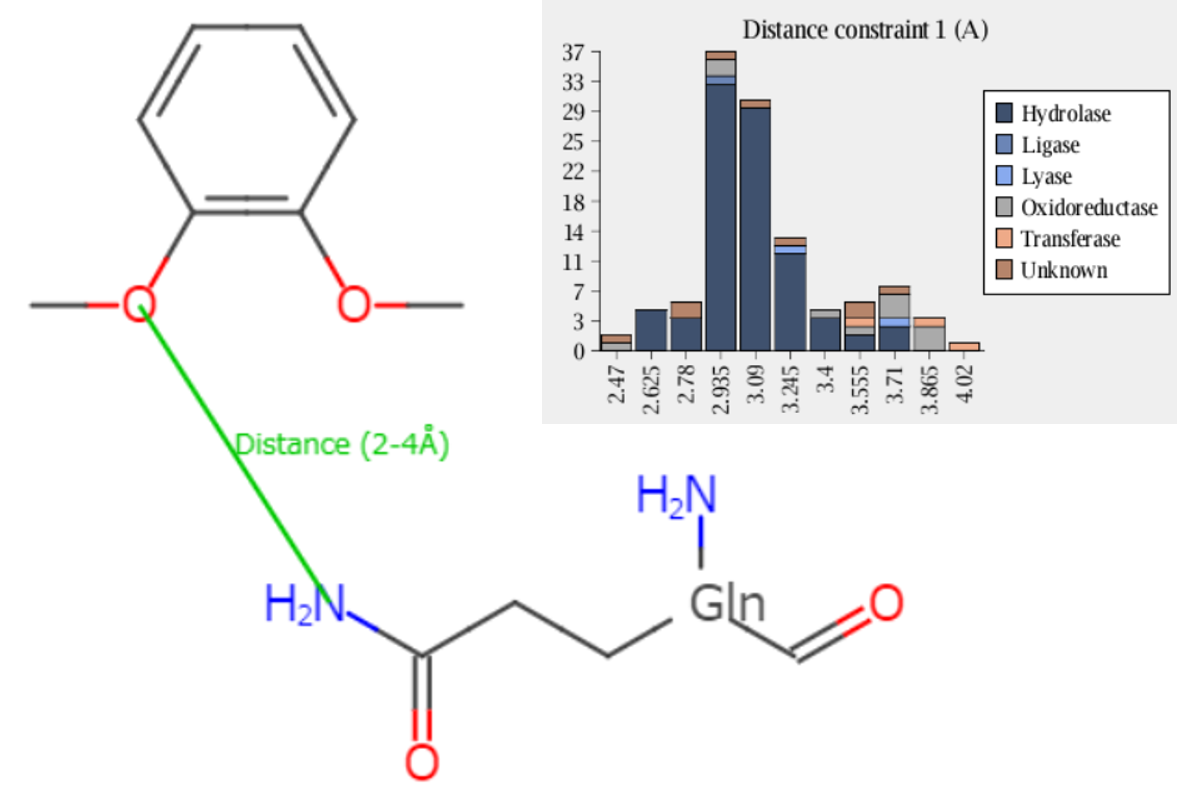
3D相互作用検索
ウェブページに埋め込まれたスケッチャーでタンパク質とリガンドの部分構造を描画して、タンパク質−リガンド間の原子間距離、角度、二面角を指定して、検索します。
化合物部分構造・類似構造検索
ウェブページに埋め込まれたスケッチャーで構造を指定し、リガンドの構造検索を行います。
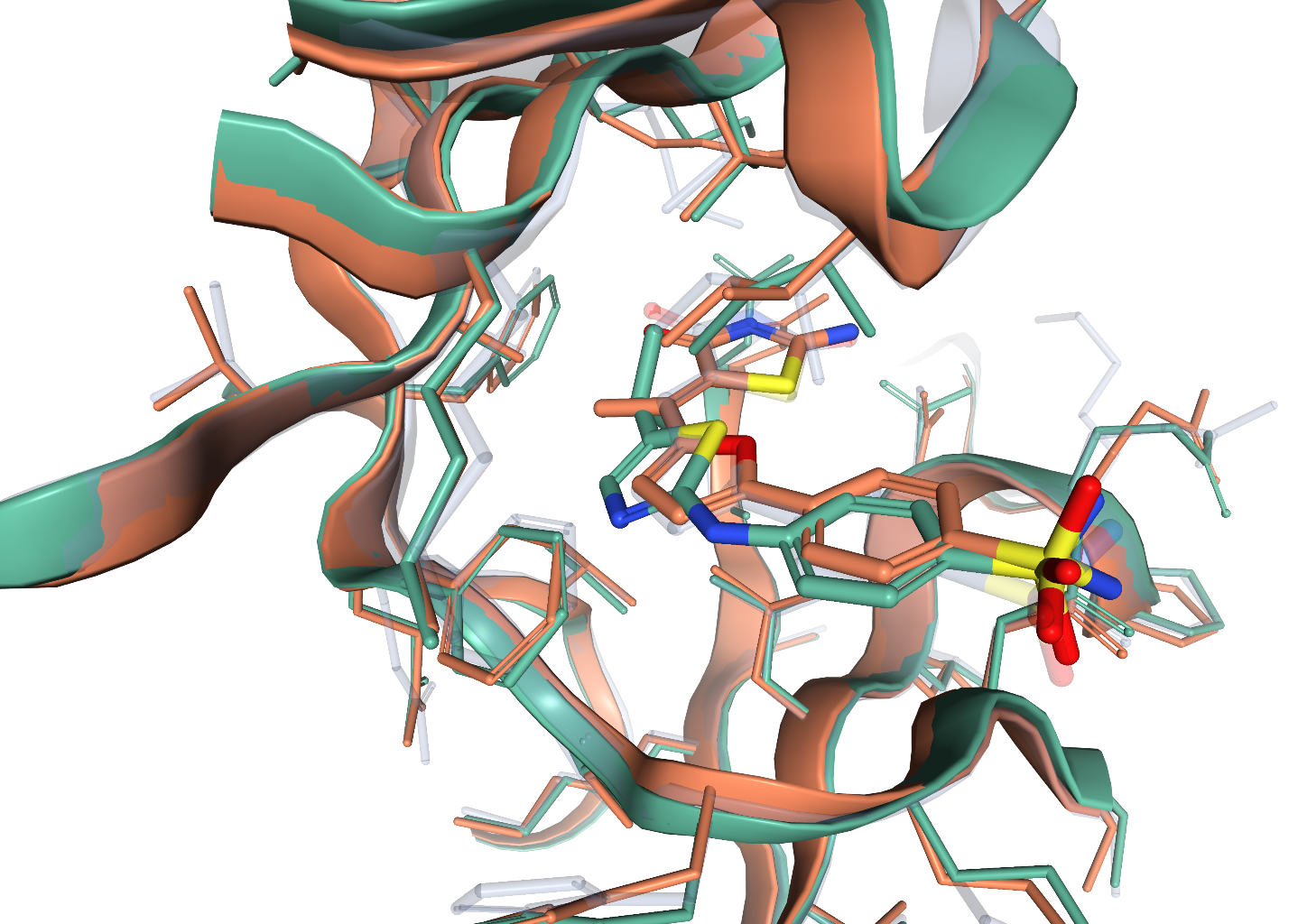
タンパク質立体構造の重ね合わせ
タンパク質立体構造全体の重ね合わせ
リガンド結合部位に重み付けした配列アラインメントを基準に重ね合わせます。
ポケット類似性による重ね合わせ
配列に依らずリガンド結合部位において、同じ性質を持つ残基が重なるように重ね合わせます。
リガンド構造による重ね合わせ
低分子の構造アラインメントによる重ね合わせを行います。
重ね合わせ構造における相互作用の比較
比較する2つの2D相互作用図を作成して、タンパク質−リガンド相互作用の違いを分かりやすく表示します。
重ね合わせ構造のMOEへの読み込み
重ね合わせ構造をMOEに取り込み、より詳しい解析を行えます。
データ表示
リガンド結合部位の2D/3D表示
リガンドごとに、タンパク質のリガンド結合部位の2D/3D図を並べて表示します。3D図では、タンパク質ファミリー特有のモチーフ、温度因子、RSCC、pLDDTスコアなどによる色付けができます。さらに2D図の残基選択が3D図と連動します。
電子密度図の描画
2Fo-Fc図などの電子密度の等値面を3D描画します。
タンパク質機能部位のアノテーション
活性部位の残基、抗体のCDR、キナーゼの各種モチーフ、GPCRの膜貫通ヘリックス領域などの残基リストを表示します。
アミノ酸配列のアノテーション
InterProScan、SCOPを用いた、GO、ファミリーなど多数の配列アノテーションを表示します。
データ更新とバージョンコントロール
PDBデータの自動更新
毎週PDBデータの更新に合わせて、自動的に最新データに更新します。個々のPDBデータの更新履歴も確認できます。
インハウスデータの簡単な登録
インハウスのX線結晶構造や、タンパク質モデル構造、ドッキングシミュレーションデータなどを登録できます。ユーザーによる個別登録や、バッチ処理による一括登録もできます。
コメントフォーラム
エントリーごとに、掲示板形式のコメント欄に構造データの修正情報など任意のコメントを書き込んで共有できます。
title:{動作環境}
OS
64bit Linux
(CentOS/RHEL 6.10, Fedora Core 10, SLES 11, Ubuntu 9.10, Mint 7以上)
CPU
Intel Xeon 3.0 GHzマルチコア 16コア 以上
RAM
1コアあたり4GB以上、合計64GB以上
ストレージ
4TB以上の空き容量。RAID, LVM, ZFSなどの冗長構成。 インハウスデータ、AlphaFoldデータを入れる場合は 相応の追加ストレージが必要。SSDを推奨。
ネットワーク環境
PDB(PDBj)サイトへのHTTPS接続
アプリケーション
MOE/batch 1トークン以上(動作にはMOEのライセンスが必要です。)
}:tab



